Keywords
Epigenetic; DNA Methylation; Histone; Environment
Introducción
Existe una molécula que guarda la información del funcionamiento de un ser vivo de generación en generación, en esta se encuentra escrito todo lo que ese ser vivo es durante su vida. Esta molécula es el ADN, el polímero que la evolución escogió para representar a la vida [1]. Muchos científicos se han preguntado cuál es el mecanismo de expresión del contenido del ADN y los estudios han llevado a concluir que en dicho proceso se encuentra lo más importante para comprender el funcionamiento celular [2], incluyendo su desarrollo, reproducción, enfermedades y muerte. Este proceso comprende reacciones químicas, entre las que hay un cúmulo de ellas que modifican el ADN sin que haya alteración de la secuencia de bases nitrogenadas [3]; esto se llama epigenética. Estas modificaciones epigenéticas crean epimarcas, favorables o no para el ser vivo, que se transmiten de generación en generación y están relacionadas con factores ambientales [4].
La epigenética es un factor determinante en la expresión fenotípica de rasgos [5], actitudes [6] e incluso enfermedades crónicas no trasmisibles (ECNT) como lo son la obesidad, diabetes mellitus tipo 2 (DM2) y las enfermedades cardiovasculares (CVD) [7], las cuales son capaces de heredarse por el linaje del individuo.
El propósito de este artículo es revisar el contenido de la literatura sobre conceptos y bases moleculares de la epigenética y su implicación en la fisiopatología de las ECNT, específicamente, DM2, obesidad y CVD.
Bases Moleculares de la Epigenética
La epigenética es capaz de regular y alterar la expresión génica y lo hace por medio de mecanismos muy complejos y variados como lo son: la metilación del ADN, la modificación de histonas, remodelación de la cromatina, entre otros [7,8].
Metilación del ADN
Al finalizar la II Guerra Mundial entre los años 1944-1945 una población del oeste holandés fue sometida a un estado de ayuno prolongado o inanición, lo cual se tradujo en una condición de mal nutrición con una baja carga proteica y calórica, esto generó un efecto sobre el epigenoma de madres que se encontraban en periodo de gestación. Las gestantes que lograron sobrevivir y posteriormente dieron a luz, se observaron rasgos específicos en sus hijos, ya que estos presentaron una predisposición a sufrir DM2, obesidad y CVD relacionadas con el estado de mal nutrición observado en las madres durante el embarazo [9,10]. Los patrones de metilación del ADN [11] es uno de los principales mecanismos para darle explicación a estos rasgos específicos que se evidenciaron sobre esta población [12].
La metilación actúa agregando grupos metilos en las bases nucleotídicas, por lo general en eucariotas ocurre en la citosina dando como producto 5-metilcitosina [12], las islas CpG normalmente se ubican en las regiones promotoras del ADN donde predominan dinucleótidos guanina y citosina enlazados por fosfatos [13]. La alteración en los patrones de metilación se relacionan con la aparición de enfermedades por cambios en la expresión génica, así la hipometilación se relaciona con el incremento de la expresión génica y la hipermetilación con la silenciación de genes [14,15]; estos procesos ocurren de manera particular y simultánea, son heredables y habitualmente estables [16,17].
La Figura 1A Ilustra la estructura de un gen que predispone a ECNT que ha sido metilado a nivel de su Isla CpG, la principal causa de la metilación es la frecuente actividad física acompañado con una alimentación balanceada, que conlleva al silenciamiento de dicho gen. Mientras que la ingesta de alimentos mayor a la necesaria acompañado de menor actividad física genera la expresión de un gen relacionado con ECNT que traduce a hipometilación Figura 1.
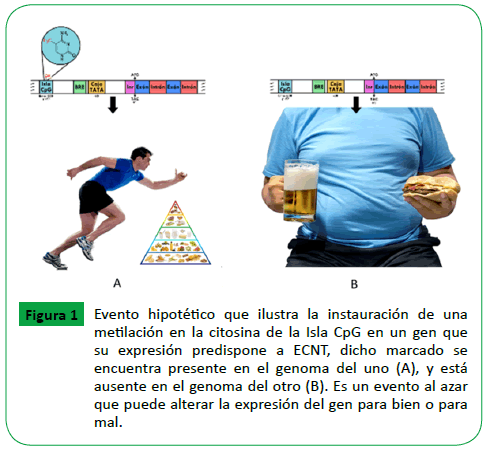
Figure 1: Evento hipotético que ilustra la instauración de una metilación en la citosina de la Isla CpG en un gen que su expresión predispone a ECNT, dicho marcado se encuentra presente en el genoma del uno (A), y está ausente en el genoma del otro (B). Es un evento al azar que puede alterar la expresión del gen para bien o para mal.
¿Cómo se mantiene la metilación?
Las epi-marcas en este caso la metilación son muy fiel respecto a la plantilla metilada, para que la replicación sea efectiva se necesita que las bases metiladas no estén en paralelo con su cadena opuesta [18,19]. Cuando ocurra la replicación solo la cadena molde se encontrará metilada y a partir de esta se origina la epimarca, con esta finalidad metiltransferasas reconocen los lugares metilados y añaden el grupo metilo (–CH3) con fidelidad a las bases citosinas que no lo están [12,20,21].
Modificación de histonas
Si bien es cierto los mecanismos epigenéticos por los cuales se modifican las histonas, llevan una estrecha relación con la estructura terciaria del ADN [22], el superenrollamiento de esta molécula permite que las aproximadas 3200X106 de pares de bases se agrupen en un lugar de apenas nanómetros [23-25].
Este gran empaquetamiento que tiene el ADN le causa un problema esencial, y es que esa información se torna inaccesible [22,26]; y ningún proceso por complejo que sea puede tener acceso a la información, es decir mientras el ADN se encuentre en su plegamiento de orden superior no será capaz ningún proceso como la replicación, transcripción, reparación, y recombinación [20,24].
Los cromosomas humanos al igual que el resto de organismos eucariotas, el ADN se encuentra supercondensado por proteínas (histonas) cuyo complejo da origen a la cromatina [27,28], las histonas son las que se encuentran en mayor medida, conteniendo principalmente arginina y lisina, aminoácidos que a pH fisiológico presentan carga positiva, manteniendo un nexo muy fuerte con las cargas negativas de los grupos fosfatos del ADN [29].
Diversos estudios han demostrado que distintos procesos en eventos post-traducionales son causantes de alteraciones de la expresión génica [30], estos procesos epigenéticos son varios, siendo la acetilación, metilación, fosforilación y monoubicuitinación de histonas ejemplos de estos [31]. Se caracterizan por alterar la arquitectura de la cromatina, lo que hace accesible al ADN a proteínas como factores de transcripción que interviene en la transcripción de genes [32,33].
El objetivo de este artículo no es tratar profundamente los procesos post-traducionales de histonas, sino utilizarlos para explicar desde la perspectiva de la epigenética la fisiopatología de las enfermedades objetivo del presente texto. Aunque es necesario exponer la acetilación y metilación de histonas, procesos que en la literatura y bases de datos presentan material suficiente para el fin último de este escrito.
Acetilación de histonas
Se ha evidenciado que en la mayoría de veces los grupos acetilo (-COCH3) se agregan o se elimina en los aminoácidos terminales de las histonas, especialmente a la lisina 9 de la histona H3, por enzimas acetiltransferasas (HATs) y por deacetilasas de histonas (HDACs), induciendo a que las pares de bases que se encuentran en esa región específica se dispersen, y se dé la transcripción génica; esto sucede si actúa una HATs. En tal sentido si actúa una HDACs eliminando el grupo acetilo induce la condensación de la cromatina deteniendo la transcripción [34,35].
Metilación de histonas
La adición de grupos metilos a los aminoácidos blancos de la metiltransferasas de histona (HMTs) provoca el cambio estructural de la cromatina [36-38]. En este caso el silenciamiento o la activación de la expresión génica va a depender del aminoácido metilado, es decir no siempre la metilación de histonas se relaciona con el silenciamiento de la transcripción [39-41]. En efecto la lisina y arginina de las histonas H3 y H4 son los frecuentes blancos de la HMTs, adquiriendo el grupo metilo que dona la S-Adenosyl methionine coenzima de la HMTs [42].
Por tanto la modificación de histonas causa alteración sobre la cromatina pero no sobre los nucleótidos [43-45].
Investigación transicional
En las últimas décadas muchos investigadores han dirigidos sus estudios para el desarrollo de tecnologías con el interés de mejorar el conocimiento de las bases moleculares de la epigenética, en efecto los resultados de estos estudios manifiestan que el ambiente en función del tiempo, genera la aparición o disminución de marcas epigenéticas [46-49]. Así lo demuestra Scott Joseph Kelly y Mark Edward Kelly dos hermanos gemelos astronautas que han viajado a la Estación Espacial Internacional (ISS), Scott Kelly tuvo una duración de 340 días a bordo de la ISS y su hermano una duración de 54 días. Los dos tienen genomas y experiencias de vida casi idénticas. Los análisis genéticos [50,51] demuestran grandes cambios provocados por el viaje espacial de larga duración; habiendo un aumento en la longitud de los telómeros y una disminución considerada en las metilaciones del ADN, estos datos corresponden a Scott frente a Mark que su duración en el espacio fue menor.
Enfermedades cardiovasculares, Obesidad, y Diabetes Mellitus Tipo 2 y su relación con las modificaciones epigenéticas.
La DM2, la obesidad y el riesgo cardiovascular son ECNT que afectan la calidad y estilo de vida de millones de individuos que las padecen; cada una de ellas se explica a partir de un complejo componente genético que va más allá de los signos y síntomas de cada una de estas patologías [45,52]; es importante mencionar que se piensa que su verdadero origen y aparición se encuentra encriptado en el epigenoma y que guarda mucha relación en el ambiente en el cual nos desarrollamos, como también con la herencia transgeneracional de marcas epigenéticas que provienen de nuestros ancestros las cuales se conservan y siguen propagándose a partir de los diversos mecanismos de la epigenética [48,53].
Evidencias del mecanismo epigenéticos en ECNT
Un equipo de genetistas de la universidad de Cambridge en el Reino Unido e investigadores de diabetes de la Universidad de Medicina de Harvard, mediante el estudio del ADN de 2 generaciones de ratones descendientes de una madre sometida a estrés durante el embarazo, hallaron grupos metilo en genes que a pesar de no estar relacionados directamente con el metabolismo, pueden jugar un rol en la aparición de la diabetes mellitus y la obesidad, incluso se pudo ver que los hijos y los nietos de la progenitora sometida a estrés prenatal se encontraban en sobrepeso y con una alta predisposición a la DM [48,49].
Cabe traer a colación un ejemplo de hermanos gemelos que comparten el mismo genoma, pero sus estilos de vida son muy diferentes, en este caso hipotético uno de los gemelos mantiene un estilo de vida saludable, se ejercita y su dieta es balanceada, mientras que el otro tiene un estilo de vida sedentario y en su dieta se evidencia un alto consumo de grasas saturadas y carbohidratos, en este caso de igual manera se observaran variaciones en el genoma [52-54].
Todas estas condiciones externas, estilos de vida y condiciones medio ambientales afectan el genoma de cada uno de estos individuos de manera distinta [52,53], por lo tanto a pesar de que comparten el mismo genoma las marcas que se instauran sobre el genoma del uno, no estarán presentes en el otro, causando que los genes de protección contra ECNT como DM y CVD se expresen en uno de ellos y estén ausentes en el otro.
Generalmente hábitos como el tabaquismo, la dieta poco balanceada y la ausencia de ejercicio diario ocasiona la instauración de marcas epigenéticas y las consecuencias de esta, en el peor de los casos contribuye a la aparición del cáncer [54].
Según Amit Khera y colaboradores demuestran como el estilo de vida influye en gran medida en el desarrollo de la enfermedad coronaria, analizaron la relación que hay entre los factores genéticos y el estilo de vida con dicha enfermedad. Un total 55.685 personas participaron en este estudio, los cuales pertenecían a diferentes razas, sexo, edad y nivel de escolarización. Se utilizó un sistema de cuatro factores para llevar a cabo la exclusión: no fumar en la actualidad, no padecer obesidad, realizar actividad física regular y mantener una dieta saludable, de manera similar, se comparó un estilo de vida adecuado, con un estilo de vida intermedia y un estilo de vida desfavorable. Los resultados de este análisis apoyan tres conclusiones notables. En primer lugar, los datos indican que la variación del ADN heredados con los factores de estilo de vida contribuye de forma independiente a una susceptibilidad a la enfermedad arterial coronaria. El estilo de vida saludable se asoció con una reducción del riesgo relativo (más del 50%) similar en las tasas de eventos a través de cada estrato de riesgo genético [55].
Existe una correlación importante entre estas tres entidades crónicas no transmisibles; la obesidad es uno de los factores de riesgo cardiovascular ya que favorece el desarrollo de dislipidemias, inflamación, estado protrombotico y la resistencia a la insulina la cual generara a largo plazo diabetes mellitus tipo 2 [56]. Esta última por si sola se considera un factor de riesgo cardiovascular mayor, ya que genera complicaciones microangiopatícas y macroangiopaticas que favorecen el desarrollo de enfermedad coronaria [57,58].
Dado a que los avances en la epigenómica han coexistido en los últimos años con la genómica [56], se han identificados genes implicados en la patogenia de las ECNT, esto ha mejorado la compresión de dichas patologías. A continuación se añaden los genes predominantes y evidencias de investigaciones de cambios epigenéticos, que brindan una comprensión en los mecanismos biológicos de la instauración de la enfermedad.
Obesidad
La obesidad es un problema de salud pública alarmante en todo el mundo [59], no solo por los problemas de salud que acarrea, sino porque supone implicaciones tanto para el riesgo de desarrollo de DM2, y CVD como también para el desarrollo de enfermedad renal crónica (ERC) [41,42]. Por añadidura los padres obesos son propensos a tener hijos con sobrepeso, para tal efecto el fenotipo de herencia de padres a hijos se desarrolla en base a los genes y el ambiente. Según Csaba P. Kovesdy e investigadores la obesidad es una epidemia mundial, y su prevalencia se incrementará en un 40% en la próxima década. En donde si no se toman las medidas preventivas necesarias no tendrá fin el desarrollo de la obesidad. Al respecto hay que resaltar que esta no es más que la acumulación de grasa corporal excesiva y resulta de la combinación de factores genéticos y ambientales [60].
Genes candidatos en la obesidad
En diversos estudios se han encontrado genes candidatos para la explicación de la obesidad en base al análisis previo de la expresión génica, entre estos encontramos una relación positiva entre la obesidad y la hipermetilación de los genes SLC6A4, CLOCK, KLF13, FTO, MC4R, BMAL1, POMC, PER2, HSB2 [61,62].
Evidencias de mecanismo epigenéticos en la fisiopatología de la obesidad
Estudios han demostrado que el estado de metilación del gen de la propiomelanocortina (POMC) en la sangre del cordón umbilical puede ser un marcador predictivo precoz de desarrollo de síndrome metabólico, autores observaron que una alta metilación en POMC estaba asociada a un menor peso al nacer y que, 7-9 años después, esos mismos niños presentaban niveles más altos de triglicéridos y de insulina en sangre [63].
Se ha observado que varios genes involucrados en el reloj circadiano tienen actividad de acetiltransferasa de histona (HATs), que se asocia a desórdenes del sueño y regulación del metabolismo lipídico hepático; y su interrupción entorpece seriamente el metabolismo normal del hígado favoreciendo así el desarrollo de la obesidad [64,65].
Un estudio importante ha identificado que la hipermetilación del gen KLF13 se relaciona con el IMC. En este se estudió el patrón cg07814318 en donde se observó que la metilación de este patrón fue mayor en las células de personas con mayor IMC que en las personas que se seleccionaron para participar como control. En el gen KLF13 se hallaron SNP´s y se cree que esas modificaciones en la secuencia dentro del gen conducen alteraciones de la metilación de las islas CpG relacionadas con la obesidad [66]. La función del KLF13 es actuar como factor de transcripción de cinc regulando la expresión de quimioquinas de células T. Se ha apreciado que la hipermetilación de este gen se ve asociado a la obesidad infantil extrema, por cada 1% de aumento en la metilación del cg07814318 se producía un aumento del índice de masa corporal y en los demás rasgos [67,68].
La obesidad predispone a diabetes e hipertensión [69,70] y tiene un impacto directo en el desarrollo de ERC y enfermedad renal crónica terminal (ERCT) [71,72]. Lo cierto es que es una enfermedad totalmente previsible y se puede prevenir a partir de la educación a la población de los riesgos de la misma y además de la implementación de un estilo de vida saludable acompañado de ejercicio.
Programación y Reprogramación de la obesidad
La transmisión de la susceptibilidad a la obesidad puede ocurrir como consecuencia de la “programación durante el desarrollo”. Esto quiere decir que el ambiente intrauterino influye en la vida fetal y neonatal [73,74], pero que así mismo influye de forma permanente en la estructura, función, y el metabolismo de los órganos principales, lo que conduce a un mayor riesgo de ECNT a lo largo del progreso de la vida [12,21]. Esto ha motivado a intervenciones nutricionales que disminuyan la programación en el embarazo y la lactancia, esta intervención suele causar cambios en la señalización de la insulina, el almacenamiento de grasas, el gasto de energía o vías de control del apetito [68]. Si bien no hay demasiados estudios en humanos; algunos han evaluado diferencias en la metilación de genes candidatos en los niños en relación a características maternas/paternas donde se han explorado los marcadores epigenéticos en la sangre del cordón umbilical en el parto y la obesidad. Estos estudios reflejan principalmente asociaciones sin aclarar, uno de los estudios analiza los perfiles de metilación de los hermanos nacidos antes y después de la cirugía de pérdida de peso materna y mostró diferencias referentes a la obesidad, la metilación de perfiles de genes implicados en la regulación de la homeostasis de la glucosa y la función inmune, algunas de estas se traducen en alteraciones de la expresión de genes y la sensibilidad a la insulina, los resultados sugieren que la pérdida de peso significativa en la madre es de importancia para el futuro de la salud cardiovascular y metabólica de la descendencia [75].
Casanello P et al, manifiesta que los estudios de metilación de genes se han centrado en los implicados en la obesidad, el control del apetito y/o el metabolismo, la señalización de insulina, la inmunidad, el crecimiento, la regulación del ciclo circadiano y genes impresos, evaluándose así su relación con una variedad de marcadores de obesidad. En conjunto, estos estudios han identificado una baja metilación del gen que codifica el factor de necrosis tumoral alfa (TNFa), la piruvatodeshidrogenasa quinasa 4 en músculo y de la leptina en sangre completa [76].
Estudios en mamíferos demuestran que la transmisión de la obesidad no solo se debe a la línea materna por un mal cuidado de la alimentación durante el embarazo, para tal efecto la alimentación del padre influye igualmente en la herencia de este padecimiento. Lo dicho anteriormente está respaldado por un estudio realizado por Anita Ost y colaboradores, los cuales trabajaron con las moscas Drosophila melanogaster, las moscas machos se sometieron a alimentos altos en azucares dos días antes de aparearse con las moscas hembras, las generaciones de estas se denominaron F1, teniendo en cuenta factores tales como: relación hombre: acoplamiento hembra, densidad de las larvas, la dieta y las condiciones ambientales. Una semana después las crías de las moscas machos se pesaron y luego se sacrificaron, un grupo de F1 el cual fue sometido a una alimentación normal, se midieron los triglicéridos y no se mostró ningún efecto en esta generación, a diferencia del otro grupo que fue expuesto a alimentos ricos en azucares, estos últimos presentaban una respuesta a la obesidad, además mostraban niveles de triglicéridos muy elevados, por lo tanto, el azúcar paternal fuera del óptimo fisiológico altera el control metabólico en el F1. Conviene subrayar que la generación F1 expuesta a una alimentación alta en azúcar no presentaba alteración en sus alas, en su crecimiento, desarrollo, sexo, o en el número de descendencia [76,77].
Por otra parte, Ost y colaboradores analizaron la posibilidad de que cambios similares en estructura de la cromatina estén asociados con la obesidad en mamíferos. El uso de conjuntos de datos de microarrays existentes obtenidos a partir de tejido adiposo de individuos delgados y de los individuos obesos, encontraron un agotamiento de las proteínas de Su (var) en tres conjuntos de datos de los seres humanos y en dos conjuntos de datos procedentes de ratones. Uno de los conjuntos de datos humanos se obtuvo a partir de muestras de tejido adiposo a partir de pares de gemelos monocigóticos, en el que uno de los gemelos era obeso y el otro era de IMC normal, esto indico que el agotamiento de la vía de Su (var) está asociado con la obesidad en individuos genéticamente iguales. Este hallazgo es coherente con la posibilidad de que el agotamiento de la vía de Su (var) puede ser provocada por causas ambientales que influyen en el genoma que está asociado con la obesidad. En definitiva permite confirmar que el estilo de vida del individuo, y el ambiente influye en sí mismo y en sus generaciones [77].
Diabetes mellitus tipo 2
La diabetes mellitus corresponde a un grupo de enfermedades metabólicas caracterizadas por una hiperglucemia resultante de defectos en la acción y/o secreción de la insulina [78].
Genes candidatos en la DM 2
Diversos estudios se ha demostrado la relación de estos genes con la aparición de la diabetes mellitus tipo 2; especialmente los genes PPARG, KNCJ11 Y TCF7L2, pero también HNF1B, WFS1, GCK, CDKN2A/2B, CDKAL1, SLC30A8, IGF2BP2, THADA, NOTCH2, CDC123, CAMK1D, HHEX, IDE, TSPAN8, JAZF1, KCNQ1, MTNR1B, ADCY5, PROX1, DGKB, ADAMTS9, IRS1, GCKR [56,79].
Evidencias de mecanismo epigenéticos en la fisiopatología de la DM2
C. Ling y colaboradores demostraron que la expresión del gen PPARGC1A y un conjunto de genes los cuales se encuentran implicados en la fosforilación oxidativa, se reduce en el músculo esquelético de pacientes con DM2 [79]. La secreción de insulina es dependiente de ATP, la producción de ATP mitocondrial necesita de la fosforilación oxidativa [77,80]. Así mismo determinaron que su expresión se altera en un 90% en los islotes de Langerhans humanos de pacientes con DM 2, debido a influencia tanto genética como epigenética, esta última se explica por un aumento doble en la metilación del ADN en el promotor del gen PPARGC1A [81].
A pesar del papel central de ATP para la secreción de insulina, la función de receptor activado gamma coactivador-1 alfa (PGC- 1α) el fenotipo del gen PPARGC1A, en los islotes pancreáticos humanos y células beta no estaba bien establecida [82].
Por último los genes relacionados con el transporte de glucosa se pueden ver afectados por las metilaciones en regiones promotoras o cualquier evento epigenético, y hacer deporte moderado-intenso se ha manifestado que provoca un efectivo cambio en los niveles de modificación de dichos genes, previniendo al organismo al desarrollo de estas enfermedades metabólicas [82,83].
Conclusiones
Se encontró una fuerte relación en la aparición de enfermedades crónicas no transmisibles por distintos eventos moleculares de la epigenética. Las epi-marcas modifican la expresión génica sin alterar la secuencia nucleótidica, estas pueden ser instauradas por diversos factores, especialmente el estilo de vida y el medio ambiente.
Lo más significativo de este evento molecular es que las marcas epigenéticas pueden ser instauradas para bien o para mal, una decisión consiente como mejorar los hábitos alimenticios y aumentar el tiempo en la práctica del deporte, puede lograr borrar estas marcas epigenéticas que no solo lo predisponen a ciertas enfermedades sino que también podrían ser trasmitidas a las siguientes generaciones. Estas razones suponen una manera de enfrentar las causas y consecuencias de las enfermedades crónicas no transmisibles donde el médico y el paciente deben ser autores de las mismas.
Recomendaciones Para Futuras Investigaciones
Se recomienda para futuras investigaciones o revisión del tema, el estudio de las bases moleculares epigenética no incluidas en el presente texto, para interpretar distintas enfermedades en especial a las ECNT. Así mismo una revisión sobre las enfermedades neurológicas y cómo influye la epigenética en el desarrollo de estas, por último enlazar la relación entre la psicología del paciente y la posible aparición de eventos epigenéticos.
25181
References
- Lodish H, Berk A, Zipursky S, Matsudaira P, Baltimore D, et al. (2000) Molecular cell biology. (4th ed.) New York: Freeman and Co.
- Portin P (2014) The birth and development of the DNA theory of inheritance: Sixty years since the discovery of the structure of DNA. J Genet 93: 293-302.
- Rico M, Vega G, Silva R, Oliva D (2014) Epigenetics, environment and asthma. Rev Alerg Mex 61: 99-109.
- Corella D, Ordovas JM (2017) Basic concepts in molecular biology related to genetics and epigenetics. Rev Esp Cardiol 70: 744-753.
- Houri-Ze’evi L, Korem Y, Sheftel H, Faigenbloom L, Toker IA, et al. (2016) A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell 165: 88-99.
- Stern PR (2013) Defeat, distress, and glucocorticoids. Sci Signal 6: 23.
- Handy DE, Castro R, Loscalzo J (2011) Epigenetic modifications. Circulation 123: 2145–2156.
- Zimmet PZ (2017) Diabetes and its drivers: The largest epidemic in human history? Clin Diabetes Endocrinol 18: 1.
- Lumey LH, Stein AD (1997) In utero exposure to famine and subsequent fertility: The dutch famine birth cohort study. Am J Public Health 87: 1962–1966.
- Huypens P, Sass S, Wu M, Dyckhoff D, Tschöp M, et al. (2016) Epigenetic germline inheritance of diet-induced obesity and insulin resistance. Nat Genet 48: 497–499.
- Rodríguez M, Téllez N, Cerbón M, López M, Cervantes A (2004) Metilación del ADN: Un fenómeno epigenético de importancia médica. Rev Invest Clín 56: 56¬71.
- Kim M, Costello J (2017) DNA methylation: An epigenetic mark of cellular memory. Exp Mol Med 49: 322.
- Zahava S, Itamar S (2010) DNA methylation and gene expression. WIREs Syst Biol Med Med 2: 362–371.
- Phillips T (2008) The role of methylation in gene expression. Nature 1: 116.
- Foret S, Kucharski R, Pittelkow Y, Lockett GA, Maleszka R (2009) Epigenetic regulation of the honey bee transcriptome: Unravelling the nature of methylated genes. BMC Genom 10: 472.
- Kaati G, Bygren LO, Pembrey M, Sjostrom J (2007) Transgenerational response to nutrition, early life circumstances and longevity. Eur J Hum Genet Genom 15: 784–790.
- Heard E, Martienssen RA (2014) Transgenerational epigenetic inheritance: Myths and mechanisms. Cell 157: 95–109.
- Mesa V, Barros P, Medina C (2006) Metilación del ADN: Marcador diagnóstico y pronóstico de cáncer. Gac Méd Méx Cell 142: 1.
- Álvarez A (2011) Evaluación del estado de metilación de genes supresores tumorales y amplificación de oncogenes, como marcadores moleculares en DNA libre en plasma de pacientes con cáncer pulmonar, atendidos en cuatro hospitales de Bogotá–Colombia. Universidad Nacional de Colombia, Bogotá D.C.
- Pierce BA (2015) Epigenética. Genética - Un enfoque conceptual. (5ª Ed.) Panamericana, Madrid, España.
- Pérez JD, Rubinstein ND, Dulac C (2016) New perspectives on genomic imprinting, an essential and multifaceted mode of epigenetic control in the developing and adult brain. Ann Rev Neurosci Cell 39: 347-384.
- Riddihough G (2015) Inheritance of a covalent histone modification. Cell 348: 87-88.
- Collins F, Morgan M, Patrinos A (2003) The human genome project: Lessons from large-scale biology. Science 300: 286-290.
- Craig J, Adams M, Myers E, Li P, Mural R, et al. (2001) The sequence of the human genome. Science 291: 1304-1351.
- Rialdi A, Campisi L, Zhao N, Cesar A, Pietzsch C, et al. (2016) Topoisomerase 1 inhibition suppresses inflammatory genes and protects from death by inflammation. Science 352: 7993.
- Moissiard G, Cokus S, Cary J, Feng S, Billi A, et al. (2012) MORC family ATPases required for heterochromatin condensation and gene silencing. Science 336: 1448-1451.
- Sánchez R, Peña P, Miska W, Henkel R (2000) Determinación de la variabilidad en la condensación de la cromatina espermática y a-glucosidasa seminal con relación al espermiograma. Rev Méd 128: 483-489.
- Ramachandran S, Henikoff S (2015) Replicating nucleosomes. Sci Adv 1: 1-9.
- McVickerG, De Geijn V, Degner JF, Cain CE, Banovich NE, et al. (2013) Identification of genetic variants that affect histone modifications in human cells. Science 342: 747-749.
- Kouzarides T (2007) Chromatin modifications and their function. Cell 128: 693-705.
- Brian MC (2012) Regulación de la expresión génica: Cómo operan los mecanismos epigenéticos. Arch Argent Pediatr 110: 132-136.
- Cedar H, Bergman Y (2009) Linking DNA methylation and histone modification: Patterns and paradigms. Nat Rev Genet 10: 295-304.
- Kumar R, Deivendran S, Santhoshkumar TR, Pillai MR (2017) Signaling coupled epigenomic regulation of gene expression. Oncogene 36: 5917-5926.
- Cheung P, Allis CD, Sassone-Corsi P (2000) Signaling to chromatin through histone modifications. Cell 103: 263-271.
- Wei W, Liu X, Chen J, Gao S, Lu L, et al. (2017) Class I histone deacetylases are major histone decrotonylases: Evidence for critical and broad function of histone crotonylation in transcription. Cell Research 27: 898-915.
- Poulard C, Bittencourt D, Wu DY, Hu Y, Gerke DS, et al. (2017) A post‐translational modification switch controls coactivator function of histone methyltransferases G9a and GLP. EMBO reports 18: 1442-1459.
- Yan H, Zhang D, Liu H, Wei Y, Lv J, et al. (2015) Chromatin modifications and genomic contexts linked to dynamic DNA methylation patterns across human cell types. Scientific Reports 5: 8410.
- Wang W, Sidoli S, Zhang W, Wang Q, Wang L, et al. (2017) Abnormal levels of histone methylation in the retinas of diabetic rats are reversed by minocycline treatment. Scientific Reports 7: 45103.
- Greer EL, Shi Y (2012) Histone methylation: A dynamic mark in health, disease and inheritance. Nature Reviews Genetics 13: 343-357.
- Kim S, Kaang BK (2017) Epigenetic regulation and chromatin remodeling in learning and memory. Experimental & Molecular Medicine 49: e281.
- Serra Valdés MA, Ruíz MS, Viera García M (2018) Las enfermedades crónicas no transmisibles: Magnitud actual y tendencias futuras. Scielo 8: 140-148.
- GBD (2015) Risk Factors Collaborators. Global, regional, and national comparative risk assessment of 79 behavioral, environmental and occupational, and metabolic risks or clusters of risks,1990–2015: A systematic analysis for the Global Burden of Disease Study 2015. Lancet 388: 1659-1724.
- Cervantes R, Presno J (2013) Fisiopatología de la diabetes y los mecanismos de muerte de las células β pancreáticas. Revista de Endocrinología y Nutrición 21: 98-106.
- González E (2013) Obesidad: Análisis etiopatogénico y fisiopatológico. Endocrinol Nutr 60: 17-24.
- Hernández A, Duque J, Rosales W, Lizcano F (2017) Perspectivas moleculares en cardiopatía hipertrófica: Abordaje epigenético desde la modificación de la cromatina. Rev Col Cardiol 24: 146-152.
- Csaba P, Furthc S, Zoccali C (2017) Obesidad y enfermedad renal: Consecuencias ocultas de la epidemia. Sociedad Española de Nefrología 30: 360-369.
- Elizabeth J, Mitsuteru R, Shi H, Corish J, Yamazawa, K, et al. (2014) In utero undernourishment perturbs the adult sperm methylome and intergenerational metabolism. Science 345: 1255903-1255908.
- Callaway E (2014) Starvation in pregnant mice marks offspring DNA. Nature 511: 1.
- Macías K, Zazueta V, Mendoza C, Rangel A, Adilla F (2008) Epigenética, más allá de la Genética. Acta Universitaria Universidad de Guanajuato 18: 50-56.
- Witze A (2017) Astronaut twin study hints at stress of space travel. Nature 541: 1.
- https://www.nasa.gov/feature/how-stressful-will-a-trip-to-mars-be-on-the-human-body-we-now-have-a-peek-into-what-the-nasa
- López P, Reya J, Gómez D, Rodríguez Y, López J (2011) Combatir la epidemia de diabetes mellitus tipo 2 en Latinoamérica: características especiales que demandan acciones innovadoras. Clin Invest Arterioscl 23: 90-99.
- Lando HA, Hipple BJ, Muramoto ML, Klein JD, Prokhorov AV, et al. (2010) El tabaco es un problema que afecta a los niños en el mundo entero. Boletín de la Organización Mundial de la Salud 88: 2.
- Fernández EM, Figueroa O (2018) Tabaquismo y su relación con las enfermedades cardiovasculares. Rev Haban Cienc Méd 17: 225-235.
- Khera A, Connor A, Emdin P, Drake I, Natarajan P, et al. (2016) Genetic risk, adherence to a healthy lifestyle, and coronary disease. N Engl J Med 375: 2349-2358.
- Wiebe JC, Wägner AM, Novoa FJ (2011) Genética de la diabetes mellitus. Nefrología Suplemento Extraordinario 2: 1-119.
- Barrès R, Yan J, Egan B, Treebak JT, Rasmussen M, et al. (2012) Acute exercise remodels promoter methylation in human skeletal muscle. Cell Metab 15: 405–411.
- Fuster V, Ibáñez B (2008) Diabetes y enfermedad cardiovascular. Rev Esp Cardiol Supl 8: 35-44.
- Menéndez R, Gómez D, Lardoeyt R (2015) Contribution of the interaction of genome and environment in the onset of type 2 diabetes mellitus. Rev Ciencias Médicas 19: 465-477.
- Oviedo G, Morón A, Santos I, Sequera S, Soufrontt G, et al. (2006) Risk factors of nontransmissible chronic diseases in students of medicine of carabobo university. Venezuela. Year 2006. Nutr Hosp 23: 288-293.
- García J, Pérez G, Beltrán J, García E, Berenguer E, et al. (2018) Biomarcadores epigenéticos: Hacia su implantación en la rutina clínica. Génetica Médica News 1: 1-23.
- Casanello P, Krause B, Castro J, Uauy R (2016) Epigenetics and obesity. Science 87: 335-342.
- Calva P, Álvarez A, Madrigal E, Espinosa E (2011) Revisión de los principales genes involucrados en el desarrollo de la obesidad. Revista mexicana de ciencias farmacéuticas 42: 26-38.
- Fermil M, Martinez J (2015) La nutrición personalizada a través de la epigenomica. Dialnet 27: 345-361.
- In-Uk K, Hye-Ja L, Joo-Yeon H, Nak-Hyun C, Suman L (2017) Obesity-related CpG Methylation (cg07814318) of Kruppel-like Factor-13 (KLF13) gene with childhood obesity. Nature 7: 45368.
- Hongkui J, Song T, Yang Y, Zhang F, Zhou Y, et al. (2015) KLF13 promotes porcine adipocyte differentiation through PPARγ activation. Cell & Bioscience 5: 5-28.
- Reig G, Concha M (2012) Impronta genómica y desarrollo embrionario. Int J Morphol 30: 1453-1457.
- Zapatero A, Barba R, Gonzalez N, loozad J, Plazae S, et al. (2012) Influence of obesity and malnutrition on acute heart failure. Rev Esp Cardiol 65: 421-426.
- López F, Cortés M (2011) Obesidad y corazón. Rev Esp Cardiol 64: 140-149.
- Guo C, Dong G, Liang X, Dong Z (2019) Epigenetic regulation in AKI and kidney repair: Mechanisms and therapeutic implications. Nat Rev Nephrol 15: 220-239.
- Sharifian R, Denisenko O, Johnson Z, Bomsztyk G (2018) Distinct patterns of transcriptional and epigenetic alterations characterize acute and chronic kidney injury. Sci Reps 8: 17870.
- Monk D, Mackay D, Eggermann T, Maher E, Riccio A (2019) Genomic imprinting disorders: Lessons on how genome, epigenome and environment interact. Nat Rev Genet 20: 235-248.
- Radford EJ (2017) Exploring the extent and scope of epigenetic inheritance. Nat Rev Endocrinol 14: 345–355.
- Casanello P, Krause B, Castro J, Rodríguez R (2016) Epigenética y obesidad. Revista Chilena de Pediatría 87: 335-342.
- Ozanne S (2015) Epigenetic signatures of obesity. New England J Med 372: 973-974.
- Öst A, Lempradl A, Casas E, Weigert M, Tiko T, Et al. (2014) Dieta paterna define descendencia estado de la cromatina y la obesidad entre generaciones. Cell 159: 1352-1364.
- Saenz de Pipaon M, Dorronsoro I, Álvarez-Cuervo L, Butte NF, Madero R, et al. (2017) The impact of intrauterine and extrauterine weight gain in premature infants on later body composition. Pediatr Res 82: 658-664.
- Juvenal GJ (2016) Epigenetic: An old Word, new concepts. Rev Argent Edocrinol Metab 51: 66-74.
- Mootha VK, Lindgren CM, Eriksson KF, Subramanian A, Sihag S, et al. (2003) PGC-1alpha-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes. Nat Genet 34: 267–273.
- Olivares J, Arellano A (2008) Bases moleculares de las acciones de la insulina. Departamento de bioquímica, centro de investigación y de estudios avanzados del IPN 27: 9-18.
- Ling, C, Del Guerra S, Lupi R, Rönn T, Granhall C, et al. (2008) Epigenetic regulation of PPARGC1A in human type 2 diabetic islets and effect on insulin secretion. Diabetologia 51: 615-622.
- Fernández T, Pirola C (2016) Epigenética y síndrome metabólico. Argent. Endocrinol 52: 35-44.
- Valladares A, Suárez F, Ana I, Burguete M (2014) Epigenética de la obesidad infantil y de la diabetes. Rev Med Inst Mex Seguro Soc 52: 88-93.