Key words
deoxyribonucleic acid (DNA), allelic frequency, Zacatecas (Mexico), short tandem repeat (STR), haplotypes, polymerase chain reaction (PCR)
Introducción
A lo largo de la historia, siempre ha existido un gran interés por conocer la identidad de los individuos. Identificar a una persona, establecer su individualidad, es determinar aquellos rasgos o conjunto de cualidades que la distinguen de todas las demás y que hacen que sea ella misma. Todos los individuos de la especie humana son idénticos entre sí en un 99.5% de su ADN y sólo en el 0.5% restante (~10 millones de nucleótidos) residen las diferencias entre unos y otros y que nos hacen seres únicos (salvo en el caso de gemelos univitelinos que son genéticamente idénticos). Dentro de esta pequeña proporción de ADN distintivo existen regiones hipervariables o polimórficas que son las que nos permiten usar la información genética con fines de identificación [1, 2, 3, 4].
La tipificación de STR del cromosoma Y ha sido una herramienta de gran importancia en análisis forenses [5]. Recientemente, la comisión de ADN de la Internacional Society for Forensic Genetics (ISFG) ha publicado guías con recomendaciones sobre el uso de polimorfismos de Y-STRs en identificación humana y análisis de parentescos [6]. De igual manera, en México [7] se ha iniciado con la elaboración de bases de datos que den soporte a los análisis forenses que se han estado llevando a cabo a nivel Judicial y Federal. Nuestro análisis es un esfuerzo para colaborar en la creación de bases de datos que apoyen los resultados emitidos de genética forense en nuestro estado.
La genética forense ha revolucionado en los últimos años gracias a las técnicas de biología molecular, en especial por el descubrimiento de las regiones hipervariables, la introducción de la reacción en cadena de la polimerasa (PCR) y la electroforesis capilar. Esto ha permitido el estudio y la medición de la variabilidad genética a nivel molecular y han supuesto una de las mayores innovaciones, especialmente en aspectos relacionados con la investigación de personas o restos cadavéricos, análisis de vestigios biológicos en criminalística e investigación biológica de paternidad [8, 9].
Los microsatélites poseen una herencia mendeliana simple, lo que significa que un individuo hereda uno de los alelos de cada progenitor. En el caso de los STRs del cromosoma Y, estos son heredados de padre a hijo (varón) mas no a la hija. Los marcadores utilizados en análisis patrilineal en regiones de no recombinación son transmitidos en bloque a los descendientes varones [10, 11].
Material y métodos
Muestras
La recolección de muestras se realizo mediante hisopado bucal y sangre utilizando tarjetas FTA ® [Whatman]. Las muestras eran secadas a temperatura ambiente, se guardaron y etiquetaron para ser llevadas al laboratorio de genética donde se guardaron en refrigeración a -20°C hasta su extracción donde se utilizó una resina quelante llamada Chelex ® [BIO-RAD].
Análisis del ADN
Se amplificaron de manera simultánea 11 locis del kit comercial PowerPlex (DYS389I, DYS390, DYS389II, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS392, DYS437, DYS438) en 89 muestras; en las 41 muestras restantes de utilizó el kit Y-filer de AppliedBiosystems amplificando 5 loci más (DYS456, DYS458, DYS635, Y GATA H4, DYS448). En la PCR para el kit Y-filer se utilizaron los siguientes reactivos: 3 μl de ADN, 4.75 μl de Buffer 10X, 2.5 μl de primer, 0.25 μl de Taq Polimerasa y como diluyente 1.5 μl de agua desionizada. Para el kit PowerPlex la concentración de los reactivos fue 1.5 μl de ADN, 1.2 μl de primer, 1.2 μl de Buffer, 0.2 μl de Taq Polimerasa y como diluyente 9.4 μl de agua desionizada estéril. Las etapas en el amplificación en el termociclador consistieron de la incubación inicial (95°C- 11min), desnaturalización (94°C-1min), alineación (61°C-1min), extensión (72°C-1min), extensión final (60°C-80min). Después de que las muestras fueron amplificadas se procede a separar el ADN utilizando un secuenciador automático ABIPrims3130 de Applied Biosystems que funciona a través de la electroforesis capilar. La muestra a analizar es preparada con 9.7 μl formamida, 0.3 μl LIZ (marcador interno) y 1 μl de muestra amplificada.
Análisis de dato
Las frecuencias alélicas y haplotípicas fueron obtenidas por conteo de alelos. La diversidad haplotípica fue calculada con la ecuación de Nei: h = n (1-SPi) / n-1, donde «n» es el total de haplotipos y «Pi» es la frecuencia alélica. También determinamos la diversidad génica de cada marcador utilizando la fórmula 1-SPi y el poder de discriminación para determinar la precisión de nuestros resultados y lo obtuvimos dividiendo el total de haplotipos diferentes entre el total de haplotipos [12, 13].
Resultados
En primer lugar se determinó las frecuencias alélicas de cada marcador y la diversidad génica en cada uno de ellos y, posteriormente, las frecuencias haplotípicas así como su diversidad genética que pueden ser observadas en las tablas 1 y 2. Los haplotipos de la población son enlistados de manera completa en la tabla 9 al final de este artículo.
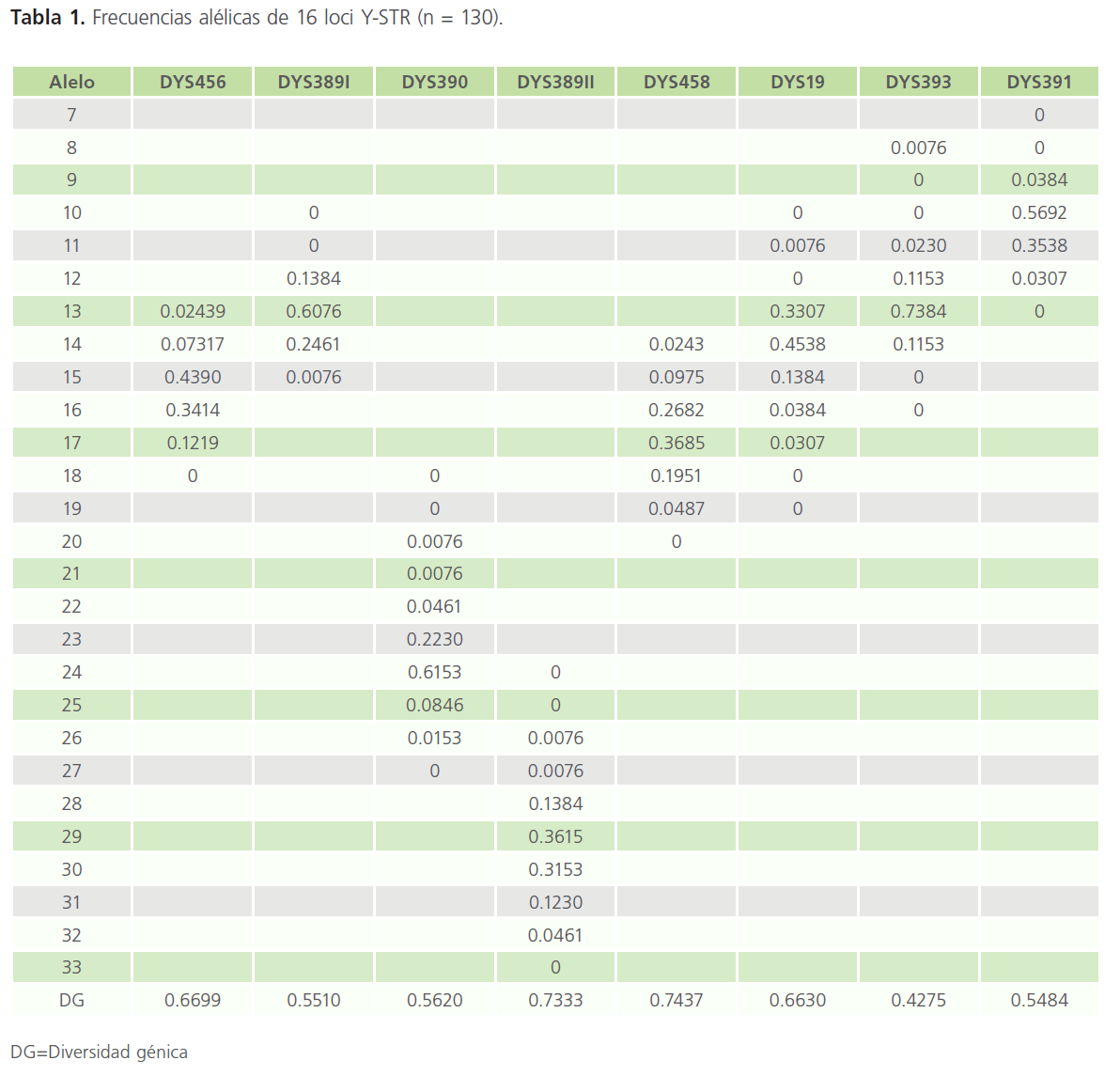
Tabla 1: Frecuencias alélicas de 16 loci Y-STR (n = 130).
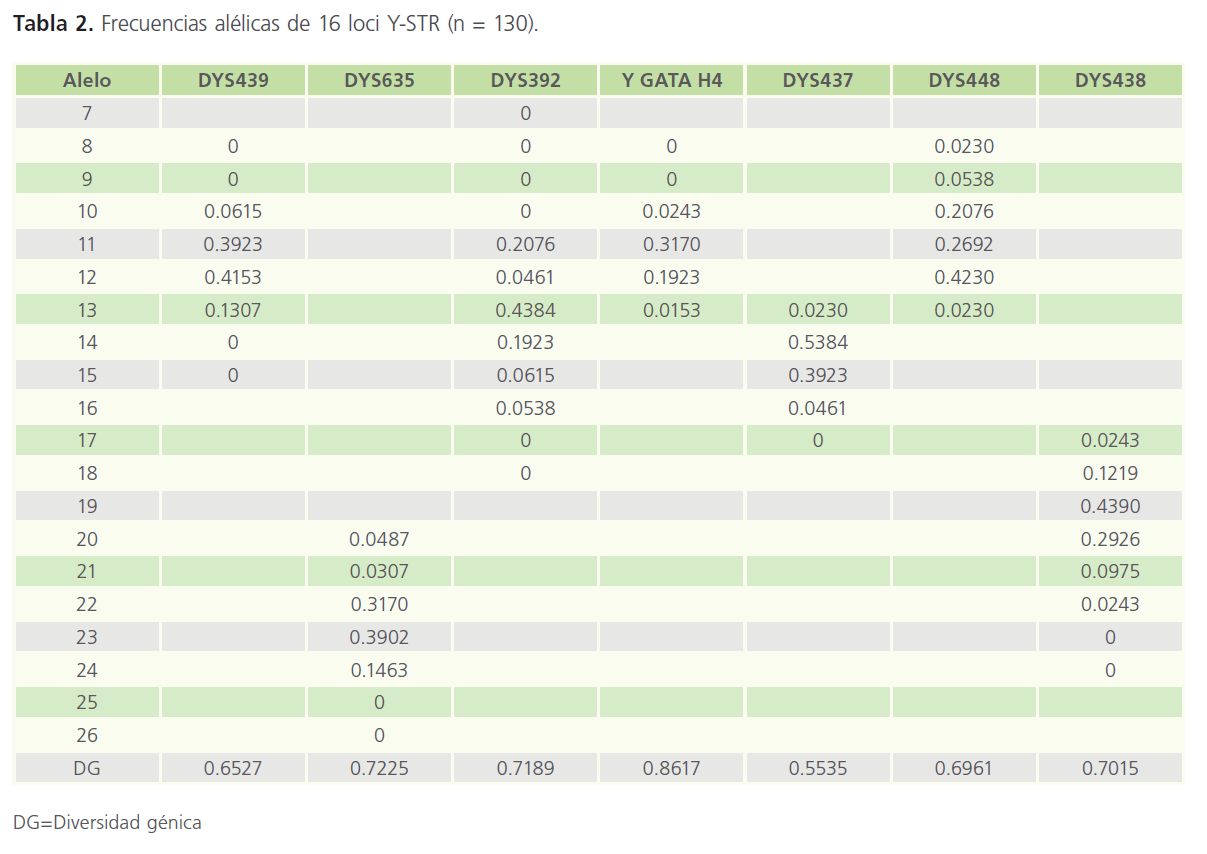
Tabla 2: Frecuencias alélicas de 16 loci Y-STR (n = 130).
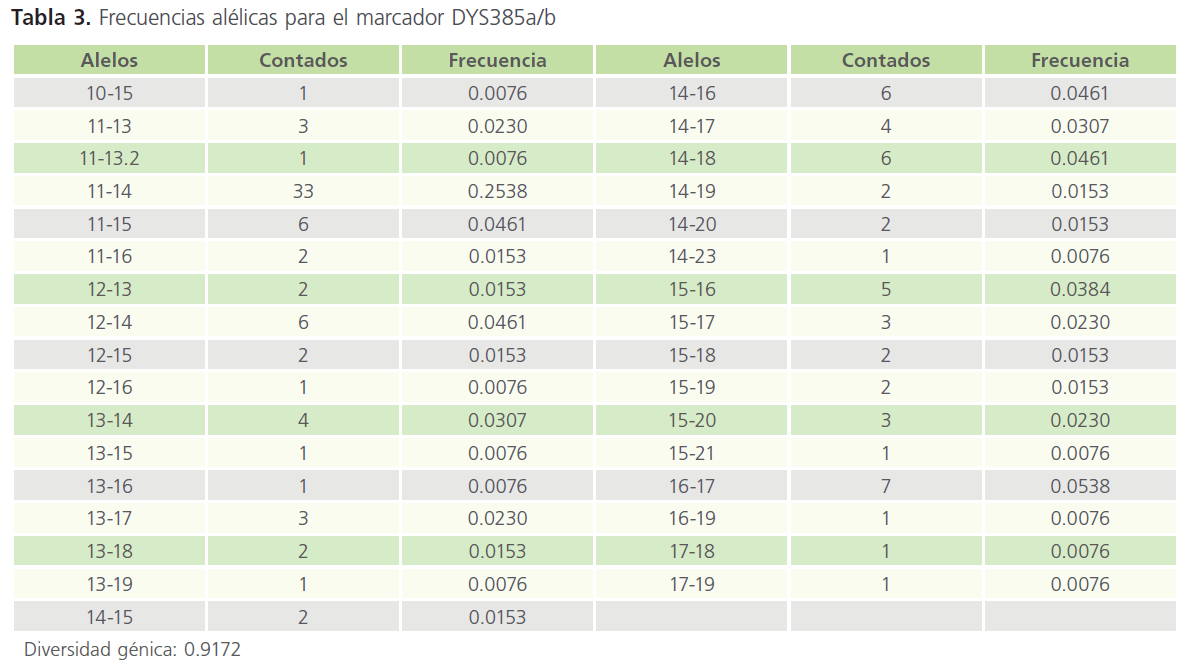
Tabla 3: Frecuencias alélicas para el marcador DYS385a/b
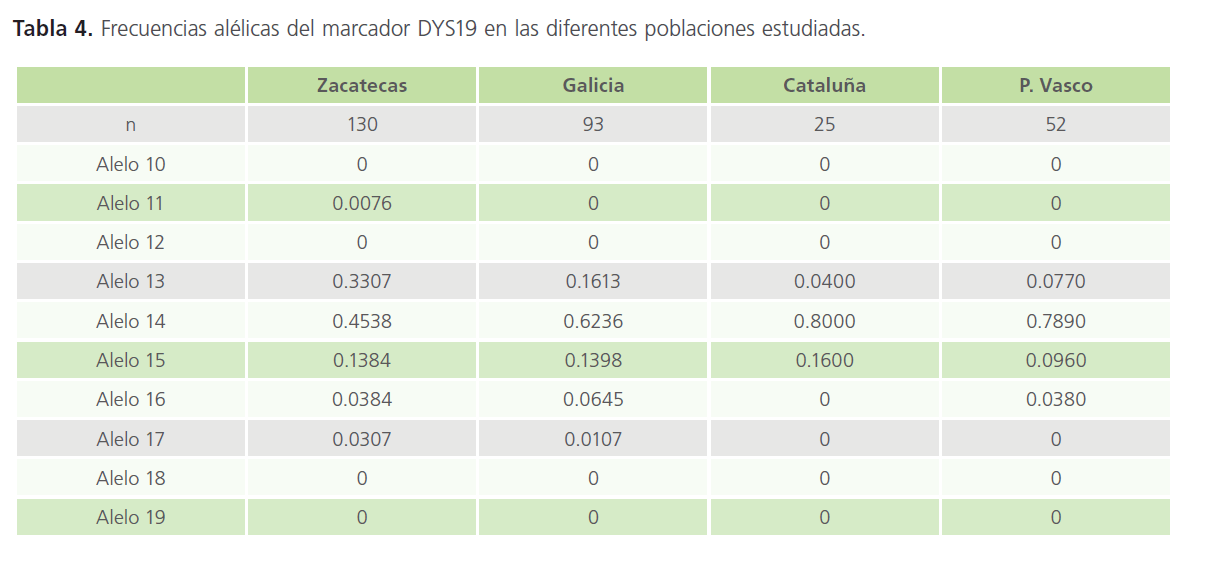
Tabla 4: Frecuencias alélicas del marcador DYS19 en las diferentes poblaciones estudiadas.
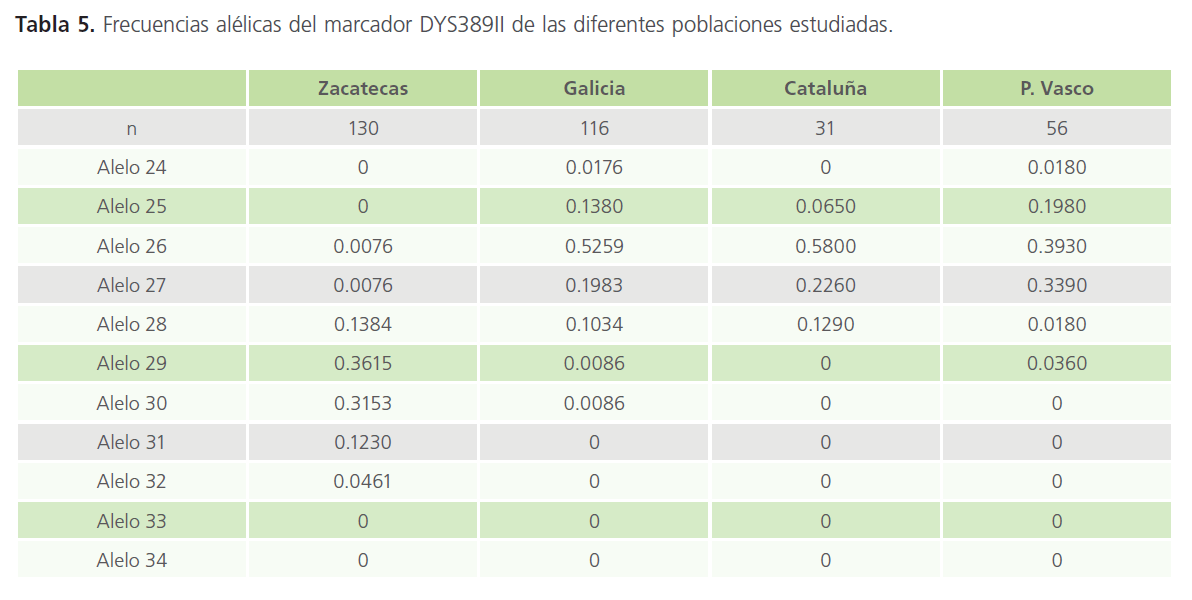
Tabla 5: Frecuencias alélicas del marcador DYS389II de las diferentes poblaciones estudiadas.
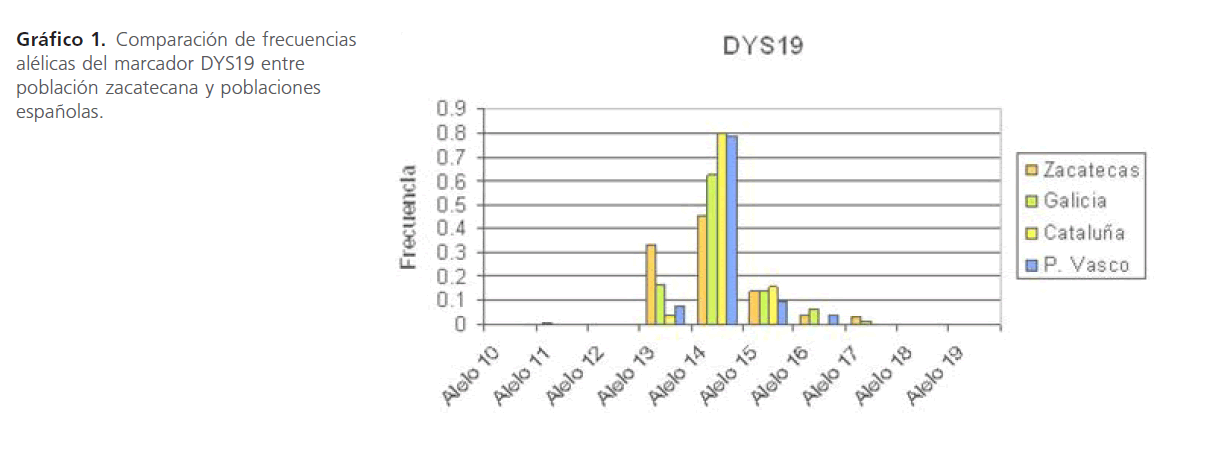
Gráfico 1: Comparación de frecuencias alélicas del marcador DYS19 entre población zacatecana y poblaciones españolas.
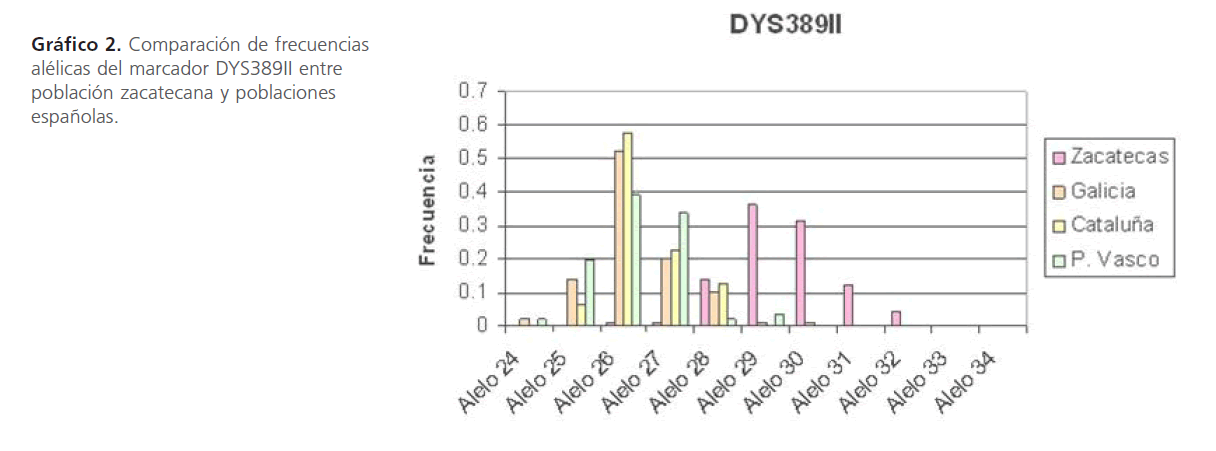
Gráfico 2: Comparación de frecuencias alélicas del marcador DYS389II entre población zacatecana y poblaciones españolas.
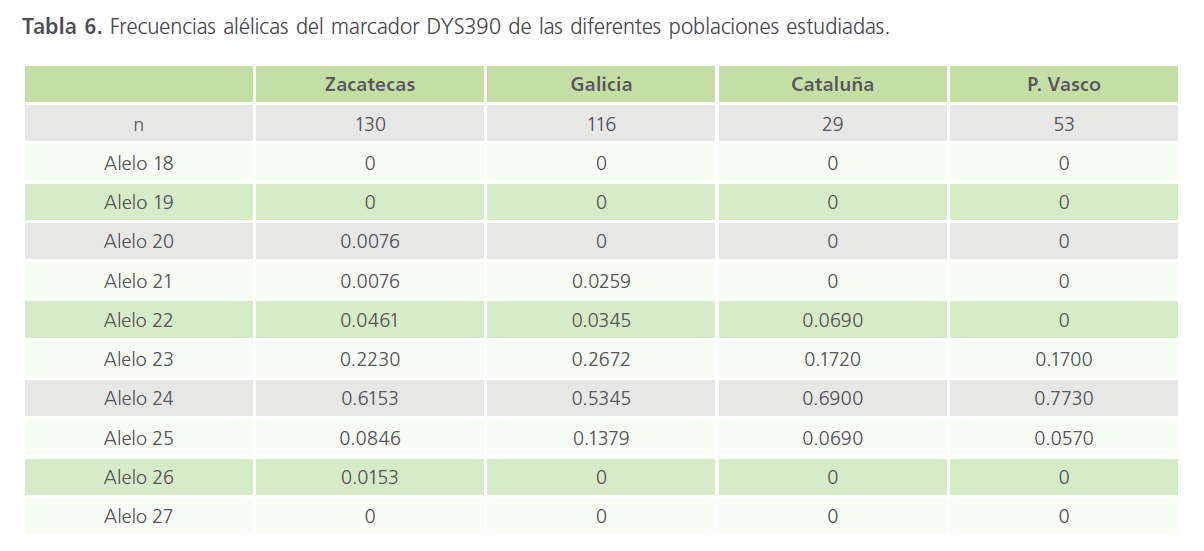
Tabla 6: Frecuencias alélicas del marcador DYS390 de las diferentes poblaciones estudiadas.
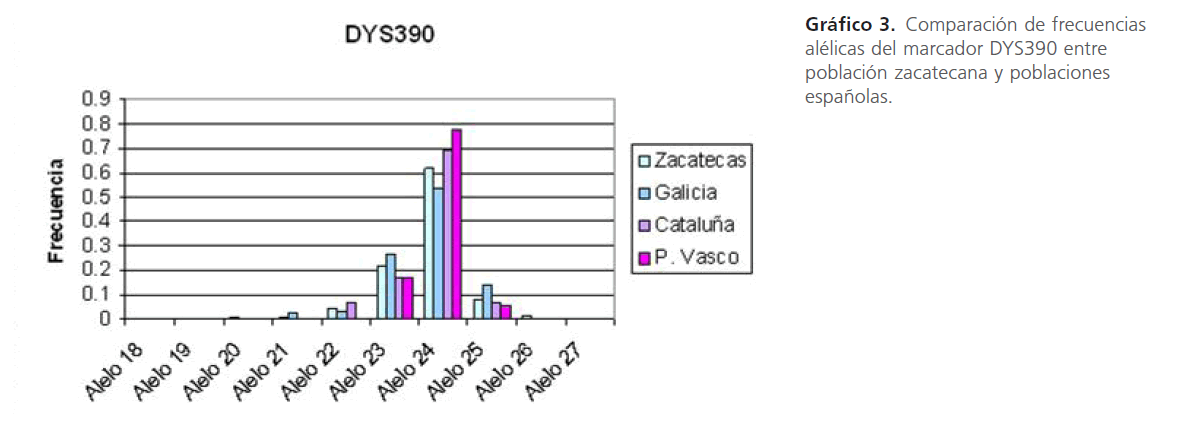
Gráfico 3: Comparación de frecuencias alélicas del marcador DYS390 entre población zacatecana y poblaciones españolas.
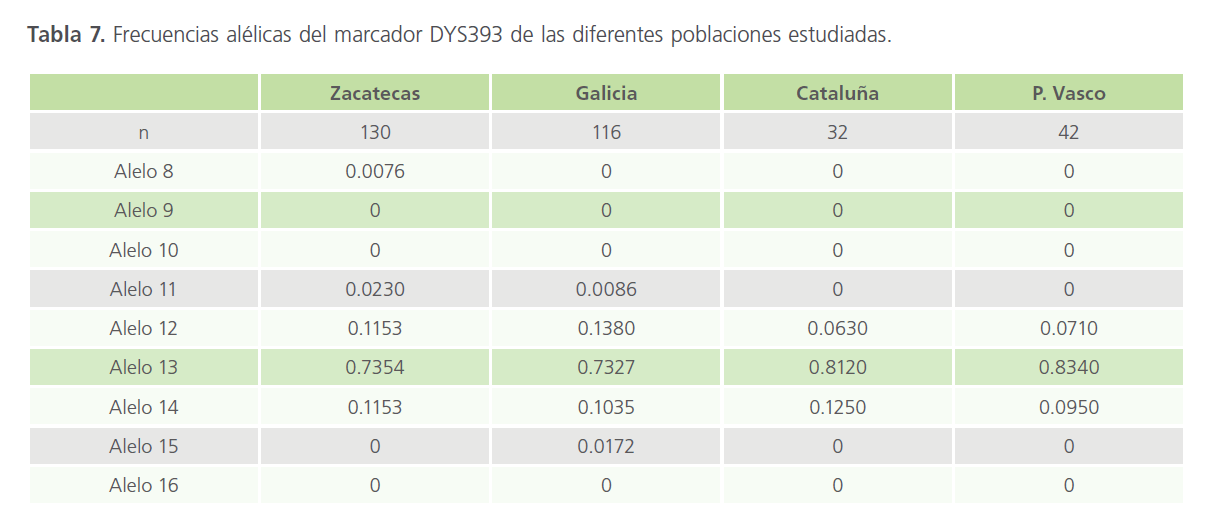
Tabla 7: Frecuencias alélicas del marcador DYS393 de las diferentes poblaciones estudiadas.
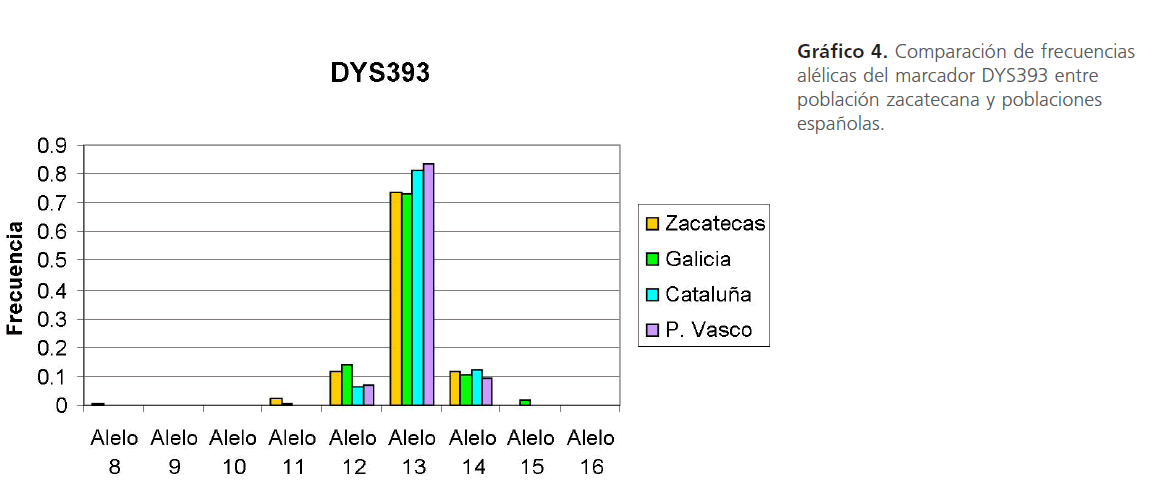
Gráfico 4: Comparación de frecuencias alélicas del marcador DYS393 entre población zacatecana y poblaciones españolas.
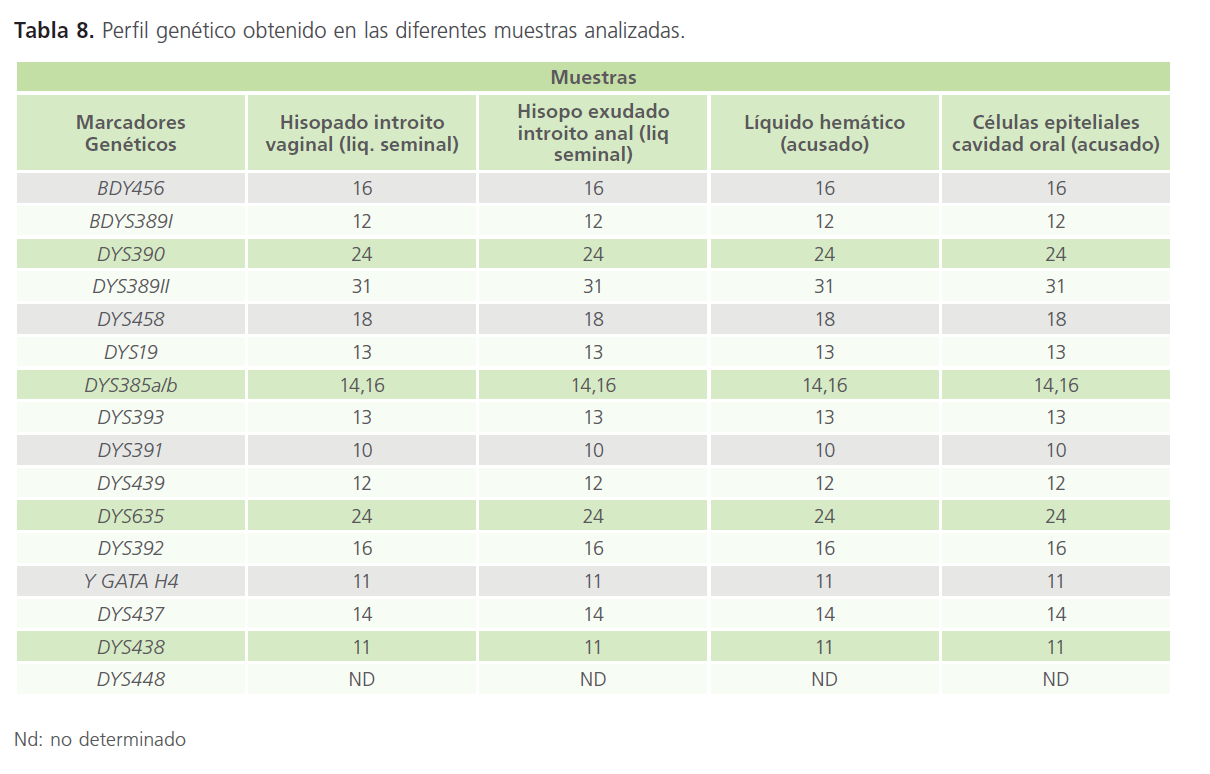
Tabla 8: Comparación Perfil genético obtenido en las diferentes muestras analizadas.
Se enlista en la tabla 3 las frecuencias alélicas del marcador DYS385a/b el cual es el que contiene una mayor diversidad génica.
De las 130 muestras analizadas se obtuvieron 127 haplotipos diferentes. El valor de diversidad génica fue mayor en el marcador DYS385 a/b con un 0.9172 (91.722%), y en cuanto a frecuencia alélica el alelo más frecuente fue el 13 del marcador DYS393 con un 0.7384 (73.84%). La diversidad haplotípica fue de 99.94%. El poder de discriminación fue 97.69%. El estudio comparativo con algunos países españoles nos muestra que no hay grandes diferencias entre las frecuencias de nuestra población con las de poblaciones españolas.
Estudio comparativo con otras poblaciones
Los resultados obtenidos en nuestra población fueron comparadas utilizando 4 marcadores (DYS19, DYS389II, DYS390, DYS393) con los datos publicados de algunas poblaciones españolas (catalana, vasca y gallega) [14].
Hemos detectado un mayor número de alelos en nuestra población, siendo el más común el alelo 14, al igual que para gallegos, catalanos y vascos. Los alelos menos representados son el 10, 18 y 19 ya que no aparecieron en ninguna de las poblaciones. Así como el alelo 11 y 17 que sólo aparecen una vez. Posiblemente la diferente distribución de alelos en nuestra población respecto a las citadas se deba al tamaño de la muestra.
Hemos encontrado 7 alelos en nuestra población. El alelo más común es el 29, mientras que en las demás poblaciones estudiadas el alelo más común es el 26.
En este marcador hemos hallado 7 alelos en nuestra población, número que se reduce para gallegos, vascos y catalanes. El más frecuente es el alelo 24, dato compartido con las demás poblaciones.
No hemos encontrado diferencias significativas para este sistema al compararlo con la población zacatecana.
Para este marcador, en nuestra población hemos observado 5 alelos, siendo más común el alelo 13 en las cuatro poblaciones estudiadas.
Discusión
Los marcadores ubicados en el cromosoma masculino resultan de gran utilidad en el establecimiento de vínculos biológicos de parentesco y en la investigación de crímenes sexuales con una obvia limitación: todos los individuos de la misma línea paterna compartirán idéntica información en estos haplotipos, por lo cual no será posible desvincular individuos relacionados por vía paterna de la posible existencia de vínculo de parentesco o filiación si sólo analizamos marcadores presentes en este cromosoma. En consecuencia, las evidencias deberán ser, en todos los casos, evaluadas también mediante marcadores autosómicos. Cuando en criminalística, por ejemplo, se analiza el ADN de una mancha de sangre encontrada en el lugar del delito y se requiere compararla con el ADN de un sospechoso es necesario conocer cual es la frecuencia en la población de los haplotipos encontrados para que, en caso de que coincidan, pueda darse un valor de probabilidad de que esa mancha de sangre procede de dicho sospechoso.
Obviamente, estudiar todos los haplotipos de una población es imposible, por lo que se toma una muestra representativa (n>100) y se analiza en ella las frecuencias de los alelos de los diferentes marcadores que serán utilizados para la identificación genética. Por tanto, las frecuencias génicas obtenidas en un estudio poblacional no son valores exactos sino estimaciones con un margen de error.
Aplicación estadística en un caso forense: a propósito de un caso
Estadística con haplotipos de Y-STR
En el caso de los marcadores Y-STR, la regla del producto basado en la multiplicación de las frecuencias alélicas a través de los marcadores no puede ser aplicada. La frecuencia del haplotipo Y-STR es comúnmente determinada por el método de conteo. Suponiendo que hay una base de datos poblacional de tamaño n con la cuenta de la base de datos x para un haplotipo particular. La frecuencia del haplotipo puede ser estimada por la proporción de la muestra, cuando x> 0, ^p=x/n,
Un intervalo de confidencia de límite superior puede ser colocado en una frecuencia de perfiles usando:

En casos donde el perfil no ha sido observado en una base de datos, el límite superior sobre el intervalo de confianza es: 1-α 1/N . Donde α es el coeficiente de coincidencia (0.05 para 95% de intervalo de confidencia) y N es el número de individuos en la base de datos.
El haplotipo obtenido de las muestras problema obtenidas de la menor y el obtenido del C. fueron verificados contra la base de datos de referencia de haplotipos del cromosoma Y en www.YHRD.org [15] y en la base de datos del haplotipo del sistema de amplificación AmpLFSTR Y Filer en www. appliedbiosystems.com/yfilerdatabase [16]. Posteriormente se determinó la frecuencia de tal haplotipo de acuerdo a la fórmula anteriormente mencionada.
Resultados
Tabla de resultados mostrando diecisiete loci STR encontrados en el cromosoma Y al ser analizadas las fracciones masculinas obtenidas de la lisis diferencial durante la extracción de ADN. Teniendo como muestra de referencia el hisopado de la cavidad oral.
Resultados obtenidos al introducir los datos en la base de datos de la compañía AppliedBiosystems.
• 7 loci haplotype (DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393): 89237 haplotypes
• 9 loci haplotype (+ DYS385a/b): 87440 haplotypes
• 11 loci haplotype (+ DYS438, DYS439): 59735 haplotypes
• 12 loci haplotype (+ DYS437): 39348 haplotypes
• 17 loci haplotype (+ DYS448, DYS456, DYS458, DYS635, YGATAH4): 27531 haplotypes.
Resultados obtenidos al comparar el haplotipo en la base de datos YHRD.
• África: Se han encontrado 0 haplotipos de 1243 en 19 poblaciones
• Oceanía / Australia: Se han encontrado 0 haplotipos de 0 en 0 poblaciones.
• Europa: Se han encontrado 0 haplotipos de 6007 en 40 poblaciones.
• Ártico: Se han encontrado 0 haplotipos de 0 en 0 poblaciones.
• Asia: Se han encontrado 0 haplotipos de 13041 en 79 poblaciones.
• America Latina: Se han encontrado 0 haplotipos de 3984 en 40 poblaciones.
• Norte America: Se han encontrado 0 haplotipos de 3256 en 4 poblaciones.
Frecuencia del haplotipo observado: El haplotipo no fue observado en las bases de datos utilizadas, sin embargo, la frecuencia para este caso en particular es de 0.19% ó 1 en 500.
Interpretación forense de los resultados
Los polimorfismos del cromosoma Y (STRs) aplicados al análisis criminalístico, presentan la ventaja de proporcionar información adicional sobre la presencia de ADN de varón en manchas forenses, particularmente en casos de violación y asalto sexual, en los que este análisis ofrece una nueva aproximación. La principal utilidad de estos sistemas se encuentra en los casos de muestras con mezclas de células masculinas y femeninas. Contrariamente a lo que sucede en los sistemas autosómicos, una elevada cantidad de ADN femenino no inhibe la amplificación de los alelos del cromosoma Y permitiendo obtener un perfil específico de ADN del asaltante.
Para interpretar el significado de una correspondencia entre muestras tipificadas genéticamente, es necesario conocer la distribución de la población de los alelos de cada locus en cuestión. Si el genotipo de la muestra de evidencia relevante es diferente del genotipo de la muestra de referencia del sospechoso, entonces el sospechoso es “excluido” como el donador de la muestra biológica estudiada. Una exclusión es independiente de la frecuencia de los dos genotipos en la población.
Si las muestras del sospechoso y la evidencia tienen el mismo genotipo, entonces el sospechoso es “incluido” como la posible fuente de la muestra de evidencia. La probabilidad de que otro individuo no relacionado corresponda a la muestra de la evidencia es estimada por la frecuencia de ese genotipo en la población relevante.
Análisis de resultados
Se realizó la comparación de las muestras en cuestión encontrando que las muestras obtenidas de la menor y las muestras de referencia del C. tienen el mismo genotipo, por lo tanto el perfil del C. está INCLUIDO como posible fuente de las muestras problema obtenidas de la menor. La probabilidad de que otro individuo no relacionado corresponda a la muestra de la evidencia es estimada por la frecuencia de ese genotipo en la población relevante, encontrando que esta es de 0.19%, es decir, la probabilidad de que otro individuo tengo el mismo haplotipo es de 1 en 500.
Se realizo el análisis estadístico de esta muestra comparándola con los datos obtenidos en nuestro estudio, y se obtuvieron los siguientes resultados:
Haplotipo
DYS456-------16
DYS389I------12
DYS390--------24
DYS389II------31
DYS458-------18
DYS19--------13
DYS385a/b----14,16
DYS393-------13
DYS391------10
DYS439------12
DYS635-----24
DYS392-----16
Y GATA H4---11
DYS437-----14
DYS438-----11
DYS448----ND
Coincidencias en nuestra base de datos
• PowerPlexY (12 loci) - 0 coincidencias en 88
• YFiler (17 loci) - 0 coincidencias en 42
Hubo 0 coincidencias en un total de 130 individuos, de los 58 municipios del estado de Zacatecas.
Cálculos de Frecuencia
En casos donde el perfil no ha sido observado en la base de datos y el intervalo de confidencia es:
α1/N 0 coincidencias en 130
1-α1/N = 1 – (0.05) [1/130] = 0.00227 = 0.227%
La principal característica que se observo al analizar todas las muestras no se amplifica el marcador DYS448 debido a que el individuo presenta una mutación en el cromosoma Y que pudo ser heredada u ocasionada por alguno de los factores mutagénicos presentados anteriormente y esto hace que tenga una mayor probabilidad de discriminación en comparación con otras muestras.
Conclusiones
PRIMERA: Se analizaron 130 muestras de las cuales se obtuvieron 127 haplotipos diferentes y se repitieron 3 haplotipos, lo que indica que en la población zacatecana sí existe diversidad de haplotipos ya que solo el 2% de las muestras se repiten y pudiera deberse a algún parentesco desconocido entre los individuos.
SEGUNDA: Los marcadores utilizados en el estudio fueron DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438, DYS448 y los valores que obtuvimos son la diversidad génica que fue mayor en el marcador DYS385 a/b con un 0.9172 (91.722%). Los alelos más frencuentes fueron el 13 del marcador DYS393 con un 0.7384 (73.84%), el 24 del marcador DYS390 con un 0.6153 (61.53%) y el 13 del marcador DYS389I con un 0.6076 (60.76%).
TERCERA: El estudio comparativo con algunas regiones españolas nos muestra que no hay grandes diferencias con nuestra población y esto puede deberse a que los mexicanos somos descendientes de los españoles y que a lo largo de los años no han habido cambios significativos en el ADN del cromosoma Y.
CUARTA: Dada la importancia que en genética forense supone la búsqueda de nuevos polimorfismos de ADN con un alto poder de discriminación y cuyo análisis sea rápido y sencillo, podemos afirmar que los loci analizados en este trabajo cumplen todos los requisitos necesarios para su aplicación en identificación humana y que son una poderosa herramienta para su en identificación humana.
Agradecimientos
Este trabajo fue particularmente apoyado por el Laboratorio de Genética Forense de la Procuraduría General de Justicia del Estado de Zacatecas, y al apoyo de la Procuraduría General de la República.
665
References
- Blanco AJ. Linajes del cromosoma Y humano; Aplicaciones genético-poblacionales y forenses. Universidad de Santiago de Compostela: Servizo de Publicacións e Intercambio Científico, 2008. Tesis
- Butler MJ. Forensic DNA Typing. 2da Edición. Burlington USA. Ed Elservier. (2005).
- Cal Teba ML. Análisis de polimorfismos del ADN microsatélite del cromosoma Y. Estudio de la población de Galicia y aplicaciones forenses. Universidad de Santiago Compostela. julio 2000. Tesis
- Cook LM. Genética de Poblaciones. Barcelona. Ediciones Omega, S.A. 1979.
- Fernández F. Análisis de 15 loci tipo short tamden repeats (STR) en la población de Paraguay para su uso en identificación forense. Departamento de Medicina Legal, Toxicología y Psiquiatría. Universidad de Granada. Marzo 2008. Tesis
- Jürgen H, Henke L, Chatthopadhyay P, Kayser M, Dülmer M, Cleef S, et al. Application of Y-chromosomal STR Haplotypes to Forensic Genetics. Croatian Med J.2001; 42(3):292-297,2001.
- Guardado M, Queipo G, Meraz-Ríos M, Berumen-Campos J. Diversidad genética en la población mexicana: Utilización de marcadores de ADN. Hospital General de México. Medigraphic Artemeni lsína. Jul-sep 2008; 71 (3):162 – 174.
- Penacino G. Análisis de ADN: 12 Marcadores del Cromosoma Masculino en la Investigación Criminal. Unidad de Análisis de ADN. Colegio Oficial de Farmacéuticos y Bioquímicos de Capital Federal, y Sociedad Latinoamericana de Genética Forense (SLAGF). Informédica J. 2004.
- Rodriguez R. La huella genética en Medicina Legal. ADN con fines forenses (Monografia de Internet). Medicina Forense y Legal. Genetica. 2007. Disponible de: https://www.portalesmedicos.com/ publicaciones/articles/643/1/La-huella-genetica-en-Medicina-Legal- ADN-con-fines-forenses.html
- Butler MJ, Kline C, Decker AD. Addressing Y-chromosome STR Allele Nomenclature. J Genetic Gen.2008; 4(2):125-148.
- Butler MJ. Genetics and Genomics of Core Short Tandem Repeat Loci Used in Human Identity Testing. J Forensic Sci. Mar 2006. 51(2).
- Gusma L, Butler MJ, Carracedo A, Gill P, Kayser K, et al.DNA Commission of the International Society of Forensic Genetics (ISFG): an update of the recommendations on the use of Y-STRs in forensic análisis. Forensic Sci Int. 2006; 157: 187–197.
- Kam Fun W, Yue-Qing H. Statistical DNA Forensics. Theory, Methods and Computation. England. Wiley. 2008
- Martínez Esther. Análisis y comparación del 16 loci STR en cromosoma Y de varios grupos poblacionales (apellidados Colón o no apellidados Colón) en la cuenca noreste mediterránea. Universidad de Granada Facultad de Medicina Departamento de Medicina Legal. Mayo 2008. Tesis
- Y Chromosome Haplotype Reference Database (YHRD). [base de datos de Internet]. Actualizado 28 mar 2012. Dsiponible en: https://www. yhrd.org/
- Applied Biosystems. YFiler Haplotype Database. [Base de datos de Internet]. Dsiponible en: https://www6.appliedbiosystems.com/ yfilerdatabase/