Introducción
La vida es un fenómeno rítmico. La actividad y funciones de los seres vivos se manifiestan como variaciones periódicas o rítmicas y no como un proceso continuo. A estas variaciones se les conoce como ritmos biológicos. En la naturaleza se observan diversos ritmos biológicos; aquellos con una periodicidad menor a 24 horas se denominan ultradianos, como son: el consumo de alimento, el ritmo cardiaco, el reposo y la actividad, etc. Por su parte, los ritmos biológicos con una periodicidad cercana a las 24 horas se conocen como circadianos, como son: el ciclo de dormir y despertar, la liberación de ciertas hormonas, etc. Mientras que los ritmos biológicos con una periodicidad mayor a un día se denominan infradianos, como es el caso del ciclo menstrual, ciclo estral, la hibernación, entre otros.
Los ritmos circadianos (del latín, circa , diano = alrededor del día), son ritmos biológicos con una duración cercana a 24 horas, autosostenibles, y que persisten inclusive en ausencia de estímulos ambientales. Estos ritmos están determinados genéticamente y son una propiedad conservada en todos los seres vivos, desde organismos unicelulares procariontes hasta mamíferos y plantas superiores. Los ritmos circadianos tienen gran importancia adaptativa, ya que modelan en el organismo, una representación del tiempo externo, para establecer una concordancia entre los sucesos del ambiente y la organización de las funciones biológicas, preparando al organismo para responder a condiciones ambientales predecibles.
En los mamíferos, los ritmos circadianos son generados por un reloj biológico central o “maestro”, localizado en unas estructuras hipotalámicas llamadas núcleos supraquiasmáticos (NSQ) en el sistema nerviosos central, y por relojes localizados en las células de diversos tejidos, conocidos como osciladores periféricos [1,2]. En conjunto, estos relojes son sistemas capaces de generar un orden temporal en las actividades del organismo, oscilan con un periodo regular y utilizan estas oscilaciones como referencia temporal interna para regular el ritmo biológico de muchas de las funciones corporales, tales como: el dormir y el despertar, el descanso y la actividad, la temperatura corporal, la presión arterial, el gasto cardiaco, el consumo de oxígeno, el equilibrio de los fluidos y la secreción de glándulas endocrinas [1,3]
Genes reloj y mecanismo molecular del reloj circadiano
Estudios de mutagénesis realizados en la mosca de la fruta (Drosophila melanogaster) permitieron identificar moscas mutantes con alteraciones del ritmo circadiano. La caracterización y análisis de estas mutantes permitió descubrir un pequeño número de genes involucrados en generar y mantener el ritmo circadiano en estos insectos. A este grupo de genes se les denominó genes reloj o “clock genes”. Posteriormente, se clonaron los genes homólogos en los mamíferos y se identificaron nuevos genes reloj a partir de hámsteres o ratones mutantes. En los mamíferos se han descrito al menos nueve genes reloj denominados: Per1, Per2, Per3, Cry1, Cry2, Clock, Bmal1, Caseína cinasa Ie (CkIe) y Rev-Erb_ [1].
El mecanismo molecular del reloj circadiano central y los osciladores circadianos periféricos, involucra la interacción de señales positivas y negativas que regulan la transcripción rítmica de los genes reloj. Este grupo de genes se encarga de controlar la actividad circadiana en todos los seres vivos [1,4]. La expresión de estos genes se regula por medio de dos asas de activación/represión transcripcional. El asa de señales positivas está controlada por los genes Clock y Bmal1, mientras que el asa negativa por los genes Per y Cry [1,5].
El modelo más aceptado para explicar el mecanismo molecular del reloj circadiano en los mamíferos es el ratón. Si situamos el funcionamiento de la maquinaria del reloj circadiano central (NSQ) del ratón en el contexto de un día circadiano de 24 horas, ocurre lo siguiente: al inicio del día circadiano (primeras horas de la mañana), la transcripción de los genes Per y Cry se activa por los heterodímeros que forman las proteínas CLOCK:BMAL1, las cuales se unen a secuencias reguladoras conocidas como cajas E, localizadas en los promotores de los genes Pery Cry para activar su transcripción. Los mensajeros de estos genes son traducidos a sus proteínas correspondientes (PER y CRY) en el citosol. Hacia la mitad del día circadiano (comienzo de la noche), las proteínas PER y CRY se asocian y forman heterodímeros (PER:CRY), que se fosforilan por la enzima CKI para después ser translocados al núcleo, en donde se acumulan. Los heterodímeros PER:CRY secuestran a los heterodímeros CLOCK:BMAL1, interfiriendo con su unión al sitio promotor de los genes Per y Cry, inhibiendo su transcripción. Por lo tanto, las proteínas PER y CRY actúan como reguladores negativos [1,5].
Los heterodímeros CLOCK:BMAL1 también activan la trascripción del gen que codifica para un receptor huérfano nuclear conocido como Rev-Erb_. Una vez en el núcleo, la proteína REV-ERB_ se une al promotor del gen Bmal1 y reprime su trascripción, provocando que los niveles del ARNm de Bmal1 caigan. Por otro lado, cuando los dímeros de las proteínas PER:CRY entran al núcleo e inhiben la trascripción de Per y Cry, también inhiben la trascripción de Rev-Erb_, e indirectamente estimulan la transcripción del gen Bmal1. En ausencia de REV-ERB_, la proteína RORa (receptor huérfano relacionado a RAR) se une al promotor del gen Bmal1 y activa su transcripción. En consecuencia, los niveles de la proteína BMAL1 aumentan al final de la noche, favoreciendo la formación de los heterodímeros CLOCK:BMAL1, los cuales activan la transcripción de los genes Per y Cry para reiniciar nuevamente el ciclo (Figura 1) [1,5].
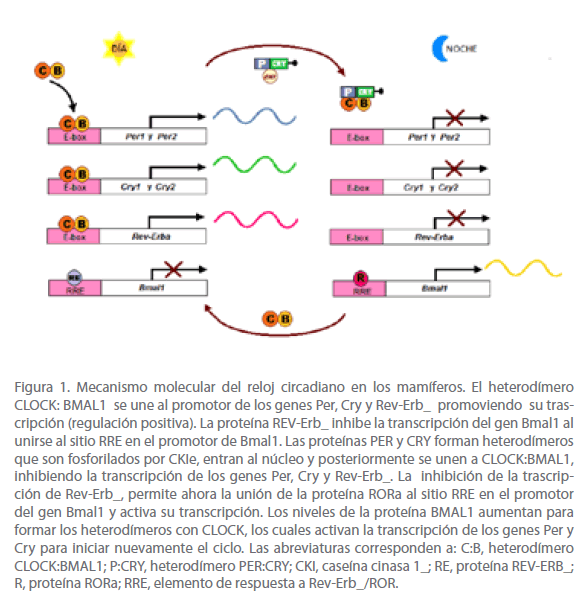
Figura 1. Mecanismo molecular del reloj circadiano en los mamíferos. El heterodímero CLOCK: BMAL1 se une al promotor de los genes Per, Cry y Rev-Erb_ promoviendo su trascripción (regulación positiva). La proteína REV-Erb_ inhibe la transcripción del gen Bmal1 al unirse al sitio RRE en el promotor de Bmal1. Las proteínas PER y CRY forman heterodímeros que son fosforilados por CKIe, entran al núcleo y posteriormente se unen a CLOCK:BMAL1, inhibiendo la transcripción de los genes Per, Cry y Rev-Erb_. La inhibición de la trascripción de Rev-Erb_, permite ahora la unión de la proteína RORa al sitio RRE en el promotor del gen Bmal1 y activa su transcripción. Los niveles de la proteína BMAL1 aumentan para formar los heterodímeros con CLOCK, los cuales activan la transcripción de los genes Per y Cry para iniciar nuevamente el ciclo. Las abreviaturas corresponden a: C:B, heterodímero CLOCK:BMAL1; P:CRY, heterodímero PER:CRY; CKI, caseína cinasa 1_; RE, proteína REV-ERB_; R, proteína RORa; RRE, elemento de respuesta a Rev-Erb_/ROR.
Los efectos opuestos de los dímeros CLOCK:BMAL1 y PER:CRY en la trascripción, aseguran que los mensajeros de Per y Cry oscilen en antifase con el mensajero de Bmal1. Los niveles máximos y mínimos en la expresión de estos genes ocurre con una periodicidad cercana a las 24 horas, de esta manera establecen el ritmo circadiano en los organismos. Por ejemplo, en el NSQ, el pico máximo de expresión de Bmal1 se da a mitad de la noche, mientras que el pico máximo de los mensajeros de Per y Cry ocurre a medio día [1].
La transmisión de la información circadiana generada a partir de la oscilación de los genes reloj en el NSQ, hacía el resto del organismo no está totalmente esclarecida. Se dice que existen dos rutas involucradas, una ruta neural y otra humoral. La primera, se genera directamente por el mecanismo molecular intrínseco del reloj, donde los heterodímeros CLOCK:BMAL1, además de participar en el control positivo de la transcripción de los genes reloj Per y Cry, actúan como factores de transcripción de otro grupo de genes que contienen cajas E en sus promotores. A estos genes se les denomina genes controlados por el reloj (clock controlled genes, CCG). Se sugiere que la actividad transcripcional de estos genes permite la transmisión de la información circadiana generada en el NSQ al resto del organismo. Un ejemplo claro de ello, es lo que ocurre con el gen que codifica para el neuropéptido vasopresina, un gen controlado por el reloj, que participa en el control circadiano de la actividad eléctrica y metabólica que caracteriza a las neuronas del NSQ. Estas neuronas, retransmiten sus señales circadianas por medio de potenciales de acción hacía otras regiones del cerebro. Son estos patrones de neurosecreción y potenciales de acción modulados en forma circadiana, los que forman el primer engranaje en la vía de salida de la información circadiana del reloj, ya que permite la comunicación entre las neuronas del NSQ y el resto de las neuronas reguladoras del Sistema Nervioso Central [6,7]. La segunda ruta, es la transmisión de la información circadiana generada en el NSQ por vía neuroendocrina, hacía el resto del organismo. Esto es posible por la comunicación que existe entre el NSQ y el eje hipotálamo-hipófisis-adrenales. Por ejemplo, la síntesis y liberación de glucocorticoides por parte de la glándula adrenal está regulada por el eje hipotálamo-hipófisis-adrenales, que a su vez recibe señales de los núcleos supraquiasmáticos por medio de la vasopresina. La secreción circadiana de glucocorticoides y otras hormonas es uno de los mecanismos de salida humorales del reloj endógeno para coordinar funciones fisiológicas, por medio de su acción activadora o inhibidora en la expresión de genes [6]. También, se ha destacado la participación del Sistema Nervioso Autónomo como un mecanismo por el cual las vías de salida del reloj transmiten la información circadiana generada en el NSQ hacía todos los órganos y tejidos periféricos [6].
Por otro lado, se ha demostrado que aproximadamente el 7% de todos los genes controlados por el reloj participan en procesos celulares importantes, como la proliferación celular o la apoptosis [8,9]. Entre estos genes se encuentran: el oncogén c-Myc, los genes supresores de tumores Trp53 y Gadd45_, así como genes que codifican para caspasas, ciclinas, factores de transcripción y los factores asociados a ubiquitinas. Otro grupo de genes que presentan patrones de expresión circadiana lo conforman: genes que tienen que ver con el metabolismo y transformación de los alimentos, por ejemplo, los genes que codifican para las enzimas: colesterol 7- hidroxilasa, PEPCK, glucógeno sintasa, glucógeno fosforilasa, y factores de transcripción que gobiernan en el metabolismo de ácidos grasos (PPAR) [10]; y genes involucrados en la destoxificación citocromo p450 y Cyp2a5 [11]. Genes relacionados con la regulación del estado redox y la regulación de la energía, como el gen de la NADH deshidrogenasa, el gen de la citocromo oxidasa y el gen que codifica para la proteína 1-del transportador de glucosa-6-fosfato [12]. Así como genes relacionados con control del citoesqueleto como la tubulina _ y tubulina b, entre otros [12]. Este podría ser también uno de los mecanismos mediante el cual el reloj circadiano controla diversas funciones celulares [8,13].
Relojes circadianos en tejidos periféricos
Existen relojes u osciladores circadianos en una gran variedad de tejidos periféricos, similares a los que operan en las neuronas del NSQ [14,15,16]. Tanto las neuronas del NSQ, como células de tejidos periféricos, y fibroblastos en cultivo muestran patrones temporales de expresión de los genes reloj. Una serie de estudios recientes han evidenciado que los genes Per, Cry y Bmal1 presentan una expresión rítmica en diversos tejidos como: hígado, corazón, pulmón, riñón, fibroblastos en cultivo y otros [1,2,15,16]. Sin embargo, a diferencia del NSQ, las oscilaciones circadianas en la expresión de los genes reloj en los tejidos periféricos de mamífero, decaen rápidamente cuando se mantienen como explantes en cultivo. En tejidos periféricos de rata, la expresión circadiana de los genes reloj se pierde en 2 a 7 días, mientras que en fibroblastos en cultivo después de 3 a 4 ciclos [15,17]. En contraste, la expresión circadiana de los genes reloj en el NSQ en cultivo se mantiene por más de un mes [17,18]. Esto ha llevado a postular que el NSQ es el reloj maestro o central con un ritmo circadiano intrínseco que sincroniza, por medio de diversas señales neurales y humorales, la actividad de los osciladores circadianos localizados en los tejidos periféricos y otras regiones del cerebro.
Por otro lado, se ha visto que diversas señales, como la administración de análogos de los glucocorticoides y el cambio en el horario de alimentación, alteran los patrones circadianos de expresión de los genes reloj en el hígado y otros tejidos periféricos, pero no afectan su expresión en el NSQ [15-17, 19, 20]. Esto sugiere que bajo ciertas condiciones metabólicas o fisiológicas, el control local del ritmo de los osciladores periféricos se puede desacoplar del control del NSQ, para adaptarse a condiciones particulares sin afectar al reloj central. Esto a su vez permitiría optimizar la fisiología celular, de tal forma que se restrinja la expresión de ciertos genes cuando sus productos son requeridos. Cuando estas condiciones particulares desaparecen, el NSQ puede sincronizar a los osciladores periféricos de regreso a su ritmo normal [2,16,17,19].
Genes reloj y cáncer
Con el fin de comprender la función de cada uno de los genes reloj en el mecanismo molecular responsable de generar los ritmos circadianos, se han desarrollado una serie de ratones mutantes o knock-out, en los que se ha inactivado de forma específica la función de estos genes. Estos ratones mutantes presentan pérdida del ritmo circadiano de actividad locomotora e ingesta de agua. Un hallazgo sorpresivo ocurrió al desarrollar ratones mutantes del gen reloj Per2, quienes además de pérdida del ritmo circadiano, desarrollaron hiperplasias de glándulas salivales, linfomas y teratomas cutáneos [13]. Al tratar a los ratones mutantes Per2 con radiaciones gamma, un agente que daña al ADN, se aceleró el desarrollo de tumores, induciendo muerte prematura en estos roedores. En contraste, este fenotipo no se observó en los ratones control (no mutantes) sometidos al mismo tratamiento [13].
Un análisis minucioso de los ratones mutantes Per2, permitió determinar que la alta incidencia de tumores se debe a una disminución en la apoptosis (muerte celular) de células dañadas. Además, se encontró que la inactivación del gen Per2 produce incremento en la expresión del oncogen c-Myc, y alteración en la expresión de los genes ciclina D1 y Gadd45_. Para explicar esta observación, estos investigadores propusieron que al inactivar el gen Per2 ocurre una disminución en la formación de los heterodímeros PER:CRY, en consecuencia menor inhibición de la transcripción del gen Rev-Erba, que se traduce en disminución en la transcripción del gen Bmal1, por lo cual los niveles del heterodímero CLOCK-BMAL1 también disminuyen. Esto es relevante, ya que el heterodímero CLOCK:BMAL1 participa en la inhibición transcripcional del oncogen c-Myc al unirse a las cajas E en su sitio promotor; por lo tanto, los niveles bajos de CLOCK:BMAL1 inducen la transcripción del gen c-Myc, provocando que sus niveles de expresión se eleven. Por su parte, los niveles elevados de c-Myc, estimulan la expresión de los genes Ciclina D1 y Gadd45_. La ciclina D1 participa en la progresión de la fase G1 a S del ciclo celular y Gadd45_ es una proteína implicada en el mantenimiento de la estabilidad genómica, reparación del ADN, control de la proliferación celular y apoptosis. Por otro lado, el heterodímero CLOCK:BMAL1 también regula la actividad transcripcional del gen Wee1, al unirse a cajas E en su promotor. La cinasa Wee1, participa en el bloqueo de la progresión celular a nivel de la fase M; por lo tanto está involucrada en la regulación negativa de la proliferación celular (Figura 2) [13]. Por otro lado, se determinó que la pérdida de función del gen Per2 en timocitos de ratón disminuye la apoptosis mediada por p53, conduciendo a la inestabilidad genómica y a la acumulación de células dañadas [13].
A partir de estas observaciones, diversos grupos de investigación en el mundo se han dado a la tarea de caracterizar la expresión de genes reloj en biopsias de diversos tipos de cáncer en humanos. En estos estudios, se ha encontrado disminución en la expresión de los genes reloj, en particular de los genes Per1 y Per2, en cáncer de mama [21,22], cáncer de pulmón [24], cáncer endometrial [25], cáncer de próstata [26], cáncer pancreático [27], entre otros (Tabla 1).
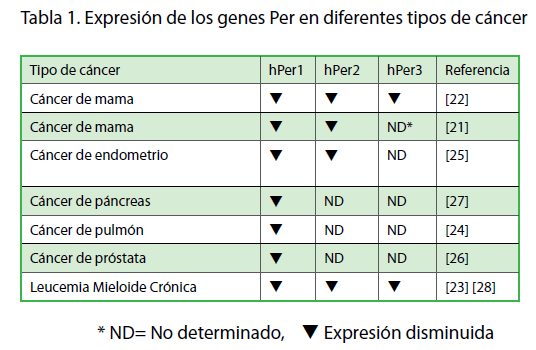
Tabla 1. Expresión de los genes Per en diferentes tipos de cáncer
Para tratar de explicar la disminución en la expresión de los genes Per en biopsias de cáncer, se han analizado los patrones de metilación de estos genes, debido a que se sabe que la metilación de los promotores, en secuencias CpG, constituye un mecanismo epigenético que regula la expresión génica. En este sentido, se encontró que los sitios de CpG en el promotor del gen hPer1 se encuentran metilados en más del 50% de los casos de cáncer de mama, lo que explica la disminución en sus niveles de expresión [22]. Un estudio similar, en pacientes con leucemia mieloide crónica, reveló que la metilación del sitio promotor de los genes hPer1, hPer2 y hPer3 correlaciona directamente con la disminución en la expresión de estos genes [23,28]. Estas evidencias sugieren que la expresión de los genes Per se encuentra controlada en parte, por mecanismos epigenéticos.
Los genes Per1 y Per2 son también supresores de tumores
La proliferación celular es un proceso esencial para la formación, reparación y mantenimiento del funcionamiento adecuado de todos los tejidos y órganos en el organismo. Los genes conocidos como supresores de tumores se encargan de orquestar los procesos implicados en el control de la proliferación y muerte celular en un organismo. Estos genes actúan como reguladores negativos de la proliferación celular, cuando no están presentes o se encuentran inactivos a causa de mutaciones, impiden que la célula prolifere normalmente, adquiriendo propiedades proliferativas características de las células tumorales.
La evidencia reciente demuestra que los genes Per1 y Per2, además de participar en el control de ritmo circadiano a nivel molecular, también actúan como genes supresores de tumores, debido a que inhiben la proliferación y estimulan la apoptosis [23,24,29-32] (Tabla 2). Se ha determinado que al sobre-expresar estos genes en diversas líneas celulares de cáncer de colon HCT116 [24], cáncer de mama MCF-7 [29] y leucemia mieloide crónica KCL22 y K562 [23], hay aumento en la apoptosis e inhibición de la proliferación celular (Tabla 2). A su vez, estos resultados correlacionan con las alteraciones en la expresión de genes clave para el control del ciclo celular (Wee1, Cdc2, ciclina D1, p21 y p53) y proliferación celular (c-Myc). El efecto de los genes Per1 y Per2 en la inhibición de la proliferación de células tumorales en cultivo se puede deber a varios eventos que incluyen, en primer lugar el bloqueo del ciclo celular, debido a alteraciones en la expresión de la ciclina D1 (proteína importante en la progresión de la fase G1-S) y alteraciones en la expresión de los genes Wee1 y Cdk1 (reguladores clave en el punto de control G2-M) (Figura 2). En segundo lugar, a la inducción en la expresión de la proteína p53, que a su vez, estimula la transcripción del gen que codifica para la proteína p21, la cual bloquea el ciclo celular en la transición G1-S, por medio de su unión a los complejos ciclina D/CDK4 y ciclina E/CDK2 (Figura 2). En tercer lugar, a la disminución en la expresión de c-Myc, que a su vez, produce una disminución en la proliferación celular. Esto debido a que c-Myc participa en la activación directa o indirecta de genes que codifican para miembros de los complejos ciclina/CDK o inhibidores de estos complejos regulando la transición G1/S del ciclo celular (Figura 2). En conjunto, estos hallazgos sugieren que los genes Per1 y Per2 participan en la supresión de tumores a través de la regulación transcripcional de algunos genes relacionados con el control del ciclo celular y proliferación celular.
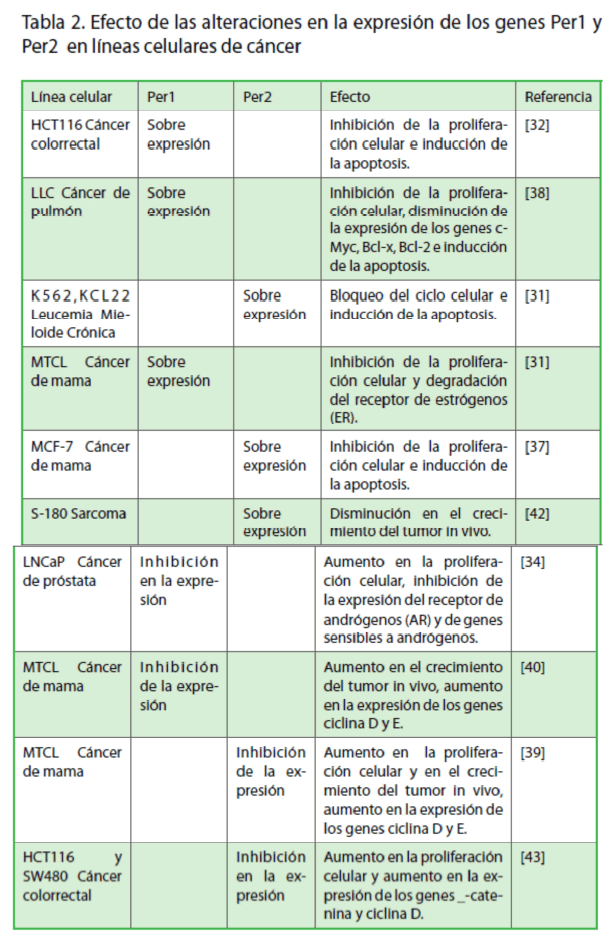
Tabla 2. Efecto de las alteraciones en la expresión de los genes Per1 y Per2 en líneas celulares de cáncer
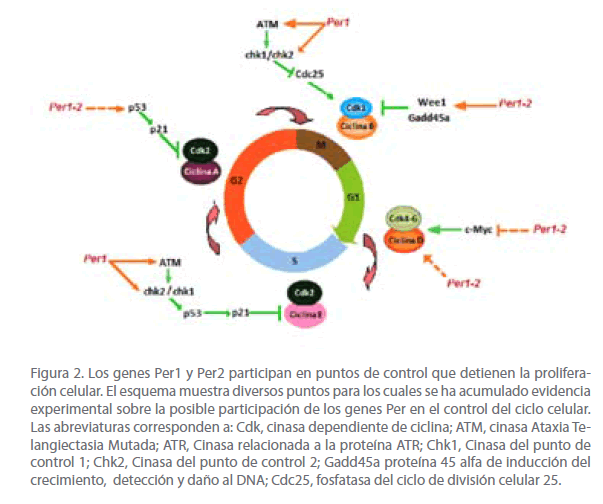
Figura 2. Los genes Per1 y Per2 participan en puntos de control que detienen la proliferación celular. El esquema muestra diversos puntos para los cuales se ha acumulado evidencia experimental sobre la posible participación de los genes Per en el control del ciclo celular. Las abreviaturas corresponden a: Cdk, cinasa dependiente de ciclina; ATM, cinasa Ataxia Telangiectasia Mutada; ATR, Cinasa relacionada a la proteína ATR; Chk1, Cinasa del punto de control 1; Chk2, Cinasa del punto de control 2; Gadd45a proteína 45 alfa de inducción del crecimiento, detección y daño al DNA; Cdc25, fosfatasa del ciclo de división celular 25.
Otros datos recientes obtenidos por Gery y col (2006), han demostrado que Per1 actúa recíprocamente con las cinasas ATM y Chk2, las cuales están involucradas en mecanismos de respuesta al daño en el ADN [24]. Estas cinasas tienen gran impacto sobre varios de los puntos de control del ciclo celular y en la respuesta contra agentes dañinos para el ADN. Cuando la cinasa ATM detecta el daño en el ADN, responde aumentando su actividad bioquímica, que modifica otras proteínas, cada una responsable de una respuesta particular en la reparación del daño al ADN, bloqueo del ciclo celular e inducción de la apoptosis.
Con la finalidad de entender de qué manera los genes Per afectan la proliferación de células cancerosas, se han realizado diversas estrategias experimentales, tanto in vivo como in vitro. Una de estas estrategias es el uso del sistema de RNA de interferencia (siRNA), que permite inhibir, por medio de pequeñas moléculas de RNA, la expresión de un gen blanco (silenciamiento génico). En un estudio realizado por Yang y col (2008) se inhibió la expresión de Per2 por medio de siRNA en líneas celulares de cáncer de mama (MTCL). En primer lugar, se observó un incremento de la proliferación celular in vitro, y un aumento en los niveles de expresión de ciclina D y ciclina E. Al transplantar las células de cáncer de mama modificadas por el sistema siRNA-Per2 en ratones, se encontró un aumento en el tamaño y la velocidad de crecimiento del tumor. Esto demuestra que el gen reloj Per2 ejerce una función supresora de tumores [31]. En estudios similares se demostró el papel supresor de tumores del gen Per1 en líneas celulares de cáncer de mama (MTCL) y cáncer de próstata (LNCap) [26,32] (Tabla 2). En conjunto, estas investigaciones sugieren que los genes Per y quizás otros genes reloj representen una nueva clase de blancos terapéuticos, cuya manipulación sería útil para modular el crecimiento y proliferación de una célula cancerosa.
Existen algunas teorías para tratar de explicar de qué manera el reloj circadiano funciona como supresor de tumores. Una de ellas fue establecida por Fu y col en el 2002, quienes propusieron que los ritmos circadianos pueden actuar como supresor de tumores debido a que participan en diversos niveles: 1) A nivel sistémico, el reloj central (NSQ) regula la proliferación celular y apoptosis en tejidos periféricos a través del sistema nervioso autónomo y sistemas neuroendocrinos, como el eje hipotálamo-pituitaria-adrenales (HPA) e hipotálamo-pituitaria-gonadal (HPG). 2) in vivo, debido a que la actividad del sistema nervioso autónomo (SNA) y de algunos sistemas neuroendocrinos está regulada por el NSQ, permitiendo una actividad rítmica de 24 horas en células de diversos tejidos periféricos. Este control se mantiene por medio de vías de señalización mediadas por proteínas G acopladas a receptores transmembranales. Por lo tanto, la alteración en los ritmos neuroendocrinos podría conducir a la desregulación del ritmo circadiano en células de tejidos periféricos y promover la oncogénesis [33]. 3) A nivel celular y molecular, debido a que los relojes circadianos regulan la expresión circadiana de diversos genes involucrados en el mantenimiento y control de la proliferación celular y la apoptosis en las células de los tejidos periféricos. Por lo tanto, debido a que estos mecanismos moleculares regulan el ciclo celular y la apoptosis, las mutaciones en los genes circadianos podrían causar la pérdida de regulación de estos procesos y el desarrollo del cáncer. En conjunto, todos estos resultados apoyan la idea de que el reloj circadiano orquesta eventos mitóticos; lo que pone de manifiesto la importancia de la regulación circadiana en las funciones celulares.
Patologías asociadas con los trastornos del ritmo circadiano El ser humano presenta una serie de cambios fisiológicos durante el transcurso del día circadiano (24 h). Por ejemplo, los cambios hormonales que ocurren al inicio del día principalmente a nivel del eje hipotálamo- hipófisis-glándulas suprarrenales, se traducen en la liberación de catecolaminas al torrente sanguíneo con su correspondiente efecto activador de las funciones orgánicas. Esto hace posible que el organismo se mantenga despierto y activo debido a un aumento en el tono muscular y en la actividad nerviosa. La liberación de catecolaminas disminuye durante la noche, con lo que disminuye también su efecto activador y se produce el sueño, periodo en el que se anulan las funciones intelectuales conscientes para dar paso a las inconscientes. Por lo tanto, la integridad de la estructura biológica circadiana es fundamental para el funcionamiento biológico y cognitivo eficiente.
Sin embargo, cuando aparecieron los trabajos en turnos rotativos, ocurrió un cambio en los hábitos conductuales del hombre, creando la necesidad de trabajar de noche y descansar durante el día. A estos cambios en los hábitos de conducta se les llama inversión del ritmo circadiano y son consecuencia de un desalineamiento del ritmo circadiano interno. Esta alteración también se ha descrito en personas que realizan vuelos intercontinentales frecuentes, sometidas a trastornos de tipo jet-lag, donde ocurre una alteración abrupta del ciclo circadiano habitual entre el sueño y la vigilia. La alteración del sistema circadiano en estas personas conlleva, a que la relación de fases normales que existen entre distintos ritmos se altere, debido a que el sistema circadiano ha sido desafiado por ciclos de luz-oscuridad inusuales. Los trastornos del ritmo circadiano ocurren como consecuencia de múltiples factores como: el jet-lag, trabajo nocturno o demoras en las fases de sueño, ronquido, obesidad, mioclono nocturno, síndrome de piernas inquietas, fobias, narcolepsia, entre otros y llegan a desencadenar una serie de patologías a largo plazo. Un ejemplo claro de ello ocurre con la pérdida del ritmo de sueño, que conduce a la aparición de enfermedades como la obesidad, envejecimiento precoz, pérdida de la memoria, etc. Por otro lado, la falta de sueño crónica está relacionada con alteraciones cardiacas, como cambios en la presión arterial (hipotensión e hipertensión) y en la frecuencia cardiaca, enfermedad coronaria e inclusive infarto de miocardio. Un grupo de investigadores afirman que existe una relación directa entre la falta de sueño y el aumento de la presión sanguínea, la cual puede derivar en ataque cardiaco y en infarto cerebrovascular [34]. Existen también otras patologías relacionadas con la pérdida del ritmo circadiano, entre las que se encuentran: problemas digestivos como gastritis, úlceras y estreñimiento, depresión, irritabilidad, trastornos neurológicos, y enfermedades metabólicas, incluyendo la diabetes. En el caso de la diabetes se sugiere que el punto de regulación de la glucosa, así como el de la insulina siguen el ritmo circadiano, por lo que se plantea que en algunos pacientes diabéticos se pierde la comunicación entre el reloj y los órganos periféricos, entre ellos el páncreas y el hígado [37].
Estudios epidemiológicos recientes sugieren que los trastornos del ritmo circadiano constituyen un factor de riesgo para el desarrollo del cáncer. En un estudio realizado con pilotos y asistentes de vuelo de diversas líneas aéreas, quienes sufren trastornos frecuentes del ritmo circadiano de tipo jet-lag, se encontró que presentan una mayor incidencia de cáncer de mama, cáncer de piel, cáncer de próstata y cáncer de colón, en comparación con la población normal [38,39]. Otros estudios epidemiológicos realizados a lo largo de varios años, han sugerido que las mujeres que trabajan de noche o que se ven sometidas a rotaciones frecuentes en el turno de trabajo muestran una mayor incidencia de cáncer de mama en comparación con las mujeres que trabajan durante el día [40-43]. En un estudio realizado por la Dra. Schernhammer y colaboradores, durante un periodo de 10 años, con la participación de más de 78000 mujeres, se evaluó la relación entre el riesgo de padecer cáncer de mama y el trabajo en turnos nocturnos. En este período se diagnosticaron 2400 casos con esta patología y mostraron una correlación con el aumento en el número de años de trabajo en turno nocturno. Estos resultados sugieren que las alteraciones de los ritmos circadianos podrían ser más importantes que la historia familiar en la determinación del riesgo a padecer cáncer de mama [40,41].
Perspectivas de la cronobiología y cronoterapia del cáncer
La cronobiología se ha establecido como una disciplina científica, cuyo campo de acción es el estudio de los eventos biológicos con relación al tiempo. La cronoterapia se define como la liberación de cantidades de un fármaco a lo largo de 24 horas, teniendo en consideración la presentación circadiana de la enfermedad o de los síntomas del proceso patológico en cuestión, con la finalidad de mejorar la eficacia terapéutica del fármaco [44]. Tomando en cuenta que varios procesos fisiológicos y metabólicos muestran fluctuaciones en el transcurso de un día, es posible administrar medicamentos en horas específicas, como se propone en el caso de la cronoterapia de fármacos anticancerígenos. En este sentido, se han realizado una serie de estudios en modelos experimentales de tumores en roedores, para evaluar la eficacia terapéutica de fármacos antitumorales administrados a diferentes horas del día. En un estudio en roedores con osteosarcoma de Glasgow se evaluó la administración de Seliciclib, un fármaco anticancerígeno que actúa como inhibidor de CDKs, a diferentes horas del día durante 5 días, demostrando que el Seliciclib reduce el crecimiento del tumor hasta 55%, en comparación con los controles que recibieron una sola dosis del fármaco [45]. Además, se encontró que la expresión de los genes reloj fue arrítmica en las células tumorales de ratones sin tratamiento; mientras que en las células tumorales de los ratones tratados con Seliciclib se recuperó cierto ritmo en su expresión [45]. Estos resultados permiten suponer que la expresión coordinada de los genes reloj en células tumorales de ratón, condujo a una mejor actividad antitumoral del Seliciclib. El reloj circadiano y sus reguladores pueden representar objetivos relevantes para el estudio de fármacos diseñados para disminuir la progresión de tumores. Otro ejemplo clásico, es la inhibición de la enzima timidilato-sintetasa, esencial para la síntesis de ADN, que representa el mecanismo de acción fundamental del fármaco antitumoral 5-Fluoruracilo (5FU). La administración de 5FU a diferentes horas del día, alrededor de 6 ciclos repetidos cada 4 horas, mostró que la toxicidad de este fármaco aumenta en la fase de actividad de los roedores [46]. Cuando el 5FU se administra a horas específicas del día en pacientes con diversos tipos de cáncer, como cáncer de colon, páncreas, pulmón u ovario, disminuye su toxicidad y mejora la tolerancia para este fármaco [47]. Sin embargo, los resultados que ofrece la cronoterapia en el tratamiento del cáncer no son inmediatos ni espectaculares, como acostumbramos observar con algunos tratamientos quirúrgicos. Pese a esto, la cronoterapia ha adquirido un creciente interés desde el punto de vista médico, ya que propone un mejor entendimiento y manipulación de la fisiología de la enfermedad, utilizando las variaciones circadianas como parte del tratamiento.
Conclusiones
Se ha demostrado que las alteraciones del ritmo circadiano constituyen un factor de riesgo para el desarrollo de diversas patologías entre las que se encuentra el cáncer, esto sugiere que la integridad del ritmo circadiano constituye un aspecto importante para la salud. También, se ha determinado que los defectos en la expresión de ciertos genes reloj, como los genes Per resultan en deficiencias en el control del ciclo celular y en la respuesta de daño al ADN, así como en el desarrollo de tumores en roedores. Es decir, el reloj circadiano no sólo organiza varios procesos biológicos y conductuales, sino también desempeña un papel fundamental en la fisiología celular. Sin embargo, son necesarios más estudios para esclarecer la participación del reloj circadiano en la proliferación celular, apoptosis y mecanismos de transformación maligna. Estas investigaciones serán de gran importancia ya que permitirán generar conocimientos valiosos para el desarrollo de nuevas opciones terapéuticas contra el cáncer.
Agradecimientos
Al Consejo Nacional de Ciencia y Tecnología (CONACYT), por la beca otorgada a Fabiola Hernández-Rosas (becario No: 223273) y a PROMEPSEP por el financiamiento otorgado a Juan Santiago-García (PTC-270).
826
References
- Reppert SM, Weaver DR. Coordination of circadian timing in mammals. Nature 2002; 418:935–41.
- Schibler U, Sassome-Corsi P. A web of circadian pacemakers. Cell 2002; 111:919-22.
- Brown SA, Schibler U. The ins and outs of circadian timekeeping. Curr Opin Genet Dev 1999; 9:588–94.
- Martinek S, Ionog AS, Manoukian Y, Young MW. A role for the segment polarity gene shaggy/GSK-3 in the drosophila circadian clock. Cell 2001; 105:769-79.
- Santiago-Garcia J, Caba M. Mecanismo molecular del reloj circadiano. En: Mario Caba, editor. Bases celulares y moleculares de los ritmos biológicos. México: Universidad Veracruzana, 2008:p.99-106.
- Hardin PE. Transcription regulation within the circadian clock: the E-box and beyond. J Biol Rhythms 2004; 19:348-60.
- Antle MC, Silver R. Orchestrating time: arrangements of the brain circadian clock. Trends Neurosci 2005; 28:145-51.
- Panda S, Antoch MP, Miller BH, Su A, Schook AB, Straume M, Schultz PG, Kay SA, Takahashi JS, Hogenesch JB. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell 2002; 109:307-20.
- Duffield GE, Best JD, Meurers BH, Bittner A, Loros JJ, Dunlap JC. Circadian programs of transcriptional activation, signaling, and protein turnover revealed by microarray analysis of mammalian cells. Curr Biol 2002; 12:551-7.
- Schibler U. Circadian rhythms. New cogwheels in the clockworks. Nature 1998; 393:620–1.
- Lavery DJ, Lopez-Molina R, Margueron R. Circadian expression of the steroid 15alpha-hydroxylase (Cyp2a4) and coumarin 7-hydroxylase (Cyp2a5) genes in mouse liver is regulated by the PAR leucine zipper transcription factor DBP. Mol Cell Biol 1999; 19:6488–99.
- Duffield GE. DNA microarray analyses of circadian timing. J Neuroendocrinol 2003; 15:991-1002.
- Fu L, Pelicano H, Liu J, Huang P, Lee CC. The circadian gene Period2 plays an important role in tumor suppression and DNA damage response in vivo. Cell 2002; 111:41-50.
- Zylka MJ, Shearman LP, Weaver DR, Reppert SM. Three period homologs in mammals: differential light responses in the suprachiasmatic circadian clock and oscillating transcripts outside of brain. Neuron 1998; 20:1103-10.
- Schibler U, Ripperger J, Brown SA. Peripheral circadian oscillators in mammals: time and food. J Biol Rhythms 2003; 18:250-60.
- Balsalobre A, Damiola F, Schibler U. A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell 1998; 93:929-37.
- Stokkan KA, Yamazaki S, Tei H, Sakaki Y, Menaker M. Entrainment of the circadian clock in the liver by feeding. Science 2001; 291:490-3.
- Abe H, T Yamashita T, Satoh H, Hoshi. Acumulation of cytoplasmic lipid droplets in bovine embryos and cryotolerance of embryos developed in different culture systems using serum-free o serum-containing media. Mol Reprod Dev 2002; 61:57-66.
- Damiola F, Minh NL, Preitner N, Kornmann B, Fleury-Olela F, Schibler U. Restricted feeding uncouples circadian oscillators in peripheral tissues from the central pacemaker in the suprachiasmatic nucleus. Genes Dev 2000; 14:2950-61.
- Yamamoto T, Nakahata Y, Soma H, Akashi M, Mamine T, Takumi T. Transcriptional oscillation of canonical clock genes in mouse peripheral tissues. BMC Mol Biol 2004; 5:1-8.
- Winter SL, Bosnoyan-Collins L, Pinnaduwage D, Andrulis IL. Expression of the circadian clock genes Per1 and Per2 in sporadic and familial breast tumors. Neoplasia 2007; 9:797-800.
- Chen ST, Choo KB, Hou MF, Yeh KT, Kuo SJ, Chang JG. Deregulated expression of the PER1, PER2 and PER3 genes in breast cancers. Carcinogenesis 2005; 1241-6.
- Gery S, Gombart AF, Yi WS, Hofmann W, Koeffler HP. Transcription profiling of C/EBP targets identifies Per2 as a gene implicated in myeloid leukemia. Blood 2005; 106:2827-35.
- Gery S, Komatsu N, Baldjyan L, Yu A, Koo D, Koeffler HP. The circadian gene Per1 plays an important role in cell growth and DNA damage control in human cancer cells. Mol Cell 2006; 22:375-82.
- Yeh KT, Yang MY, Liu TC, Chen JC, Chan WL, Lin SF, Chang JG. Abnormal expression of period 1 (PER1) in endometrial carcinoma. J Pathol 2009; 206: 111–20.
- Cao Q, Gery S, Dashti A, Yin D, Zhou Y, Gu J, Koeffler HP. A role for the clock gene Per1 in prostate cancer. Cancer Res 2009; 7619-25.
- Pogue-Geile KL, Lyons-Weiler J, Whitcomb DC. Molecular overlap of fly circadian rhythms and human pancreatic cancer. Cancer Lett 2006; 243:55-7.
- Yang MY, Chang JG, Lin PM, Tang KP, Chen YH, Lin HY, Liu TC, Hsiao HH, Liu YC, Lin SF. Downregulation of circadian clock genes in chronic myeloid leukemia: Alternative methylation pattern of hPER3. Cancer Sci 2006; 97:1298-1307.
- Xiang S, Coffelt SB, Mao L, Yuan L, Cheng Q, Hill SM. Period-2: a tumor suppressor gene in breast cancer. J Circadian Rhythms 2008; 11:4-13.
- Hua H, Wang Y, Wan C, Liu Y, Zhu B, Yang C, Wang X, Wang Z, Halberg F. Circadian gene mPer2 overexpression induces cancer cell apoptosis. Cancer Sci 2006; 97:589–96.
- Yang X, Wood PA, Oh EY, Du-Quiton J, Ansell CM, Hrushesky WJ. Down regulation of circadian clock gene Period 2 accelerates breast cancer growth by altering its daily growth rhythm. Breast Cancer Res Treat 2008; 117:423-31.
- Yang X, Wood PA, Ansell CM, Quiton DF, Oh EY, Du-Quiton J, Hrushesky W. The circadian clock gene Per1 suppresses cancer cell proliferation and tumor growth at specific times of day. Chronobiol 2009; 26:1323–39.
- Abo T, Kawamura T, Watanabe H. Physiological responses of extrathymic T cells in the liver. Immunol Rev 2000; 174:135-49.
- Martino TA, Tata N, Belsham DD, Chalmers J, Straume M, Lee P, Pribiag H, Khaper N, Liu PP, Dawood F. Disturbed diurnal rhythm alters gene expression and exacerbates cardiovascular disease with rescue by resynchronization. Hypertension 2007; 49:1104-13.
- Miyazaki K, Wakabayashi M, Hara Y, Ishida N. Tumor growth suppression in vivo by overexpression of the circadian component, PER2. Genes Cells 2010; 15:351-8.
- Wood PA, Yang X, Taber A, Oh EY, Ansell C, Ayers SE, Al-Assaad Z, Carnevale K, Berger FG, Peña M, Hrushesky W. Period2 Mutation accelerates ApcMin/+ tumorigenesis. Mol Cancer Res 2008; 6:1786–93.
- Kreier F, Buijs RM. Evidence for parasympathetic innervation of white adipose tissue, clearing up some vagaries. Am J Physiol Regul Integr Comp Physiol 2006; 293: 548–9.
- Rafnsson V, Hrafnkelsson J, Tulinius H. Incidence of cancer among commercial airline pilots. Occup Environ Med 2000; 57:175-9.
- Rafnsson V, Tulinius H, Jonasson JG, Hrafnkelsson J. Risk of breast cancer in female flight attendants: a population-based study (Iceland). Cancer Causes Control 2001; 12:95-101.
- Schernhammer ES, Laden F, Speizer FE, Willett WC, Hunter DJ, Kawachi I, Colditz GA. Rotating night shifts and risk of breast cancer in women participating in the nurses’ health study. J Natl Cancer Inst 2001; 93:1563-8.
- Schernhammer ES, Holly JM, Hunter DJ, Pollak MN, Hankinson SE. Insulinlike growth factor-I, its binding proteins (IGFBP-1 and IGFBP-3), and growth hormone and breast cancer risk in The Nurses Health Study II. Endocr Relat Cancer 2006; 13:583-92.
- Hansen J. Light at night, shiftwork, and breast cancer risk. J Natl Cancer Inst 2001; 93:1513-5.
- Davis S, Mirick DK, Stevens RG. Night shift work, light at night, and risk of breast cancer. J Natl Cancer Inst 2001; 93:1557-62.
- McMoore E. Circadian timekeeping in health an disease. Part 2 clinical implications of circadian rhythmicity. New Engl J Med 1983; 309:530-6.
- Iurisci I, Filipski E, Reinhardt J, Bach S, Gianella-Borradori A, Iacobelli E, Meijer L, Levi F. Improved tumor control through circadian clock induction by seliciclib, a cyclin-dependent cinase inhibitor. Cancer Res 2006; 66:10720-8.
- Focan C. Circadian rhythms and cancer chemotherapy. Pharmac Ther 1995; 67:1-52.
- Levi F, Granda TG. Tumor-based rhythms of anticancer efficacy in experimental models. Chronobiol Int 2002; 19:21-41.